Installation RTools

Helge Jentsch | Johannes Weidinger | Niels Schwab
RLab - Skriptbasierte modulare Umweltstatistik (Universitätskolleg 2.0)
Universität Hamburg
RLab-Impressum
Gefördert im Rahmen des „Lehrlabors“ im Universitätskolleg 2.0 aus Mitteln des BMBF (01PL17033)

Dieses Digitale Skript von Helge Jentsch, Johannes Weidinger und Niels Schwab, Universitätskolleg 2.0 / Lehrlabor, Universität Hamburg, ist lizenziert unter einer Creative Commons Namensnennung - Weitergabe unter gleichen Bedingungen 4.0 International Lizenz.
elearn.js Template
Universität Hamburg
Das elearn.js Template von Universität Hamburg ist lizenziert unter einer Creative Commons Namensnennung - Weitergabe unter gleichen Bedingungen 4.0 International Lizenz
Installation RTools
Helge Jentsch | Johannes Weidinger | Niels Schwab
RLab - Skriptbasierte modulare Umweltstatistik (Universitätskolleg 2.0)
Universität Hamburg
CC BY-SA 4.0 | 2022 - 2023
Ziel
In diesem Digitalen Skript erfährst Du, wie Du RTools auf Deinem Windows-Rechner installierst. Die Installation wird im ,,Standard''-Verlauf demonstriert.
Falls Du einen MacOS- oder Linux-Rechner benutzt, brauchst Du RTools nicht installieren.
Was sind die RTools?
RTools40 benötigst Du, um auf einem Windows-Rechner besondere, nicht-kompilierte Funktionspakete für Dein R zu installieren. Beispiele dafür sind die Pakete stringi und jsonlite.
Standardmäßig installiert R für Windows die vorkompilierten Funktionspakete von CRAN. Für diese Package-Installationen benötigst Du RTools nicht. Es handelt sich um sogenannte Binärpakete. Einige Pakete, wie die genannten, sind aber nicht vorkompiliert verfügbar. Und es gibt vorkompilierte Pakete, die von nicht-kompilierten abhängig sind, letztere werden beim Installieren automatisch mit installiert. Die nicht vorkompliereten Pakete müssen aus dem Quellcode erstellt (= kompiliert) werden.
,,Im Eifer des Gefechts'' kommt es leicht vor, dass die eher ausnahmesweise Notwendigkeit von RTools übersehen wird. Daher kann es sinnvoll sein, sie vorsichtshalber zu installieren, unabhängig davon, ob nicht-kompilierte R-Packages tatsächlich verwendet werden bzw. andere Pakete von ihnen abhängig sind. So lassen sich scheinbar unerklärliche Fehlermeldungen vermeiden.
Kompilieren
Ein in einer Programmiersprache geschriebenes Programm wird als Quellcode bezeichnet. Damit ein Programm auf dem Rechner ausgeführt werden kann, muss der Quellcode in die sogenannte ,,Maschinensprache'' übersetzt werden. Dieses Übersetzen wird Kompilieren genannt. Es wird durch einen Compiler erledigt. In RTools sind Werkzeuge enthalten, die nacheineander genutzt werden, um Quellcode zu kompilieren. Damit stellt die Toolchain der RTools eine Art Complier dar. Beim Installieren eines Packages wird durch die RTools der Quellcode der betreffenden Packages in entsprechenden Maschinencode umgewandelt, sodass auch nicht-kompilierte Packages genutzt werden können.
Laut RTools40 FAQ werden Rtools nur benötigt, wenn R-Pakete aus Quellcode kompiliert werden sollen, der ganz oder teilweise in den Sprachen C, C++ und/oder Fortran verfasst wurde.
Verwaltung der RTools
Die RTools werden momentan von Jeroen Ooms verwaltet.
Voraussetzungen
Um dieses Skript zu verstehen und die beschriebenen Schritte gleich mitmachen zu können, erfüllst Du im Idealfall folgende Voraussetzungen:
- Du hast Administrator-Rechte auf Deinem Windows-Rechner.
- Du besitzt grundlegende Kenntnisse im Umgang mit R. Am besten hast Du Dich schon mit RStudio und R bekanntgemacht.
- Du hast beide Programme installiert.
- Die R-Version sollte mindestens 4.2.x und 64-bit sein. Du bist auf der sicheren Seite, wenn Deine R-Installation aus dem April 2022 oder jünger ist. Die RStudio-Version sollte mindestens 1.2.5042 sein. Du bist auf der sicheren Seite, wenn Deine RStudio-Installation aus dem Jahr 2020 oder jünger ist.
- Starte RStudio, das mit R verbunden sein sollte.
Für Anregungen und Kommentare zur Verbesserung dieses Digitalen Skripts ist das RLab-Team immer dankbar! Du kannst auch Fragen zu den Inhalten stellen! Nutze für all das gerne die Kommentar-Funktion!
Herunterladen der Installationsdatei
Damit Du mit der Installation beginnen kannst, benötigst Du die Installationsdatei. Um diese herunterzuladen, besuche die Website von RTools. Wähle dort die aktuellste R-release-Version bzw. die zu Deiner R-Version passende.
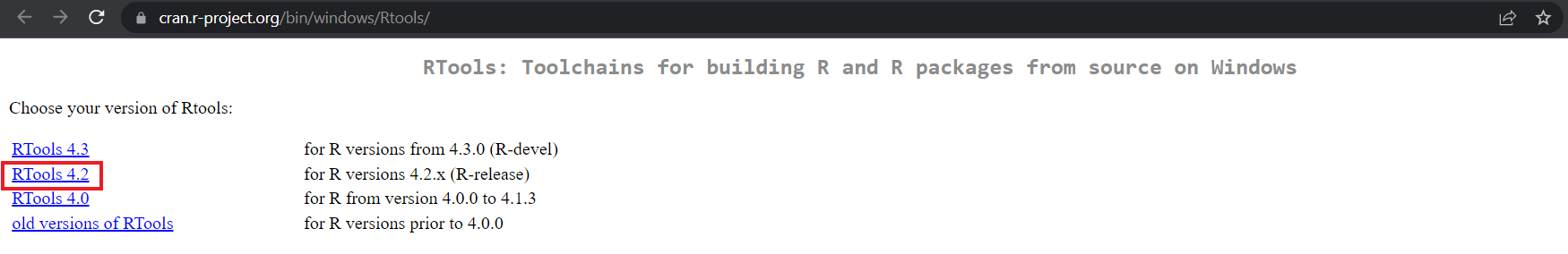
Lade auf der folgenden Seite die Datei des ,,Rtools installer" herunter und speichere sie an einem Ort, den Du wiederfindest, z.B. Deinen Standard-Downloadordner. Die Datei ist ca. 400 - 500 MB groß.

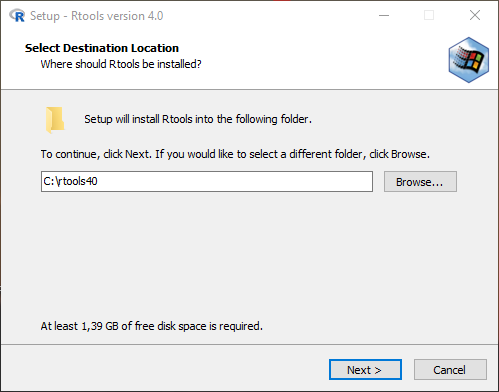
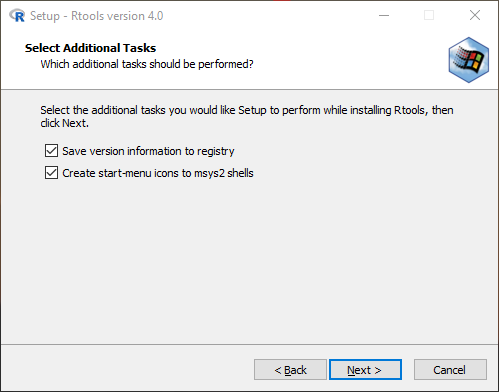
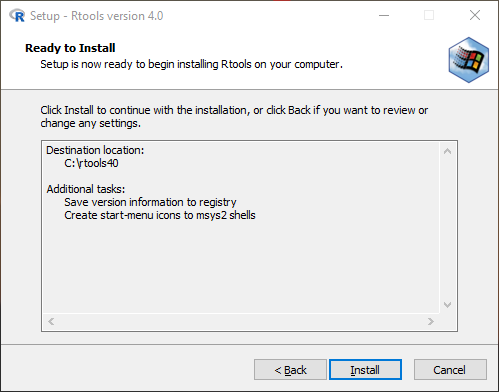
Installation
Verbindung mit RStudio herstellen
Die nächsten Schritte sind zwingend notwendig, damit Dein R auch auf die RTools zugreifen und sie zur Kompilierung von nicht-kompilierten Packages nutzen kann.
- Du hast die Installation von RTools wie im vorherigen Kapitel durchgeführt und auf Deiner Festplatte unter C:\ installiert.
- Du öffnest RStudio.
- Du schreibst (oder kopierst) in die Konsole:
writeLines('PATH="${RTOOLS40_HOME}\\usr\\bin;${PATH}"', con = "~/.Renviron")Das wird auch noch einmal in der folgenden Abbildung gezeigt:
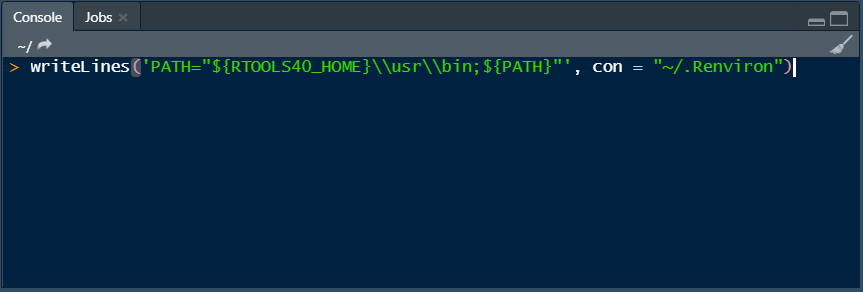
Wenn Du die Eingabe dann mit Enter bestätigst, solltest Du keinen Output in der Konsole angezeigt bekommen.
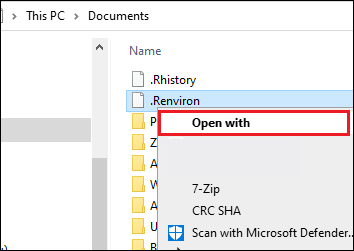
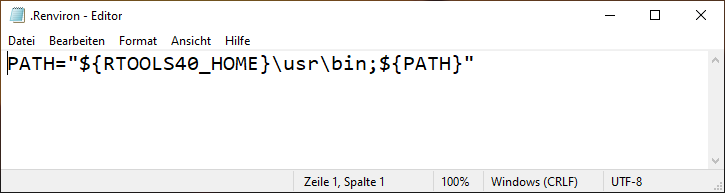
- In Deinem Dokumente-Ordner sollte nun eine neue Datei zu finden sein. Sie müsste ".Renviron" heißen. Über das Kontextmenü kannst Du sie mit dem Texteditor (z.B. Notepad / Editor) öffnen, um sie zu prüfen.
- Du siehst dann folgenden Inhalt:
- Nun hast Du die Voraussetzungen für die Verbindung erstellt. Um die Verbindung zu aktivieren, musst Du RStudio nun einmal vollständig schließen. Falls Du Skripte oder andere Dateien in RStudio geöffnet hast, solltest Du diese vorher unter einem geeigneten Namen speichern.
- Nachdem Du RStudio geschlossen hast, öffnest Du es wieder, um den vorerst letzten Schritt durchzuführen.
- In die Konsole gibst Du dazu folgendes ein und bestätigst mit einem Enter:
Sys.which("make")
Deine Ausgabe in der Konsole sollte so aussehen:
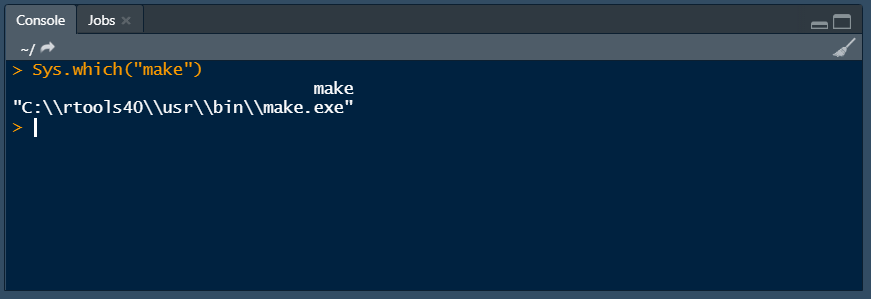
Wenn der letzte Schritt bei Dir nicht die gleichen Ergebnisse zeigt, starte RStudio noch einmal neu, überprüfe den 5. Schritt und starte bei abweichenden Ergebnissen noch einmal ab dem 3. Schritt.
Herzlichen Glückwunsch! Damit hast Du die Installation von RTools abgeschlossen und kannst nun auch Zusatzpakete, die Kompilierung benötigen, installieren. Auf der nächsten und letzten Seite dieses Digitalen Skripts erfährst Du, wie Du die Installation nicht-kompilierter R-Packages ausprobieren kannst.
Auch auf der Website von RTools40 gibt es eine (englischsprachige) Anleitung, die in den meisten Fällen für eine Standardinstallation reicht.
Zum Abschluss: Test der Installation
Ob die Installation funktioniert hat, kannst Du zum Beispiel mit einem nicht-kompilierten Package ausprobieren, das am Institut für Geographie der Uni Hamburg entwickelt wurde. Mit
install.packages("https://gitlab.rrz.uni-hamburg.de/helgejentsch/climdatdownloadr/-/archive/master/climdatdownloadr-master.tar.gz", repos = NULL, type = "source")lädst Du das Package herunter und kompilierst es direkt auf Deinem Rechner.
Alternativ kannst Du Deine Installation auch mit
install.packages('stringi')überprüfen.
Vielen Dank für die Nutzung dieses Digitalen Skripts! Für Anregungen und Kommentare zur Verbesserung dieses Digitalen Skripts ist das RLab-Team immer dankbar! Du kannst auch Fragen zu den Inhalten stellen! Nutze für all das gerne die Kommentar-Funktion!