Tipps und Tricks in R

Maria Bobrowski, Niels Schwab
RLab - Skriptbasierte modulare Umweltstatistik (Universitätskolleg 2.0)
Universität Hamburg
RLab-Impressum
Gefördert im Rahmen des „Lehrlabors“ im Universitätskolleg 2.0 aus Mitteln des BMBF (01PL17033)

Dieses Digitale Skript von Maria Bobrowski und Niels Schwab, Universitätskolleg 2.0 / Lehrlabor, Universität Hamburg, ist lizenziert unter einer Creative Commons Namensnennung - Weitergabe unter gleichen Bedingungen 4.0 International Lizenz.
elearn.js Template
Universität Hamburg
Das elearn.js Template von Universität Hamburg ist lizenziert unter einer Creative Commons Namensnennung - Weitergabe unter gleichen Bedingungen 4.0 International Lizenz
Ziel
Um die extrem steile Lernkurve zu überwinden, sind auf den folgenden Seiten einige Hinweise, Tipps und Tricks zusammengestellt, die Dir das Lernen von R erleichtern sollen:
- Vergleich zwischen GUI und IDE
- Updates von R, R-Packages und RStudio
- Abspeichern von Ergebnissen aus dem Environment und aus der Console
- Abspeichern von Plots
- Tipps und Tricks zum Abspeichern von Tabellen
- Die Kunst des Skriptens
- Beispiel R-Skript
- Kommentar-Funktion
- R Cheat Sheets

Während des Erlernens von neuen Fähigkeiten wirst Du früher oder später mit Herausforderungen konfrontiert sein. Ebenso verhält es sich beim Erlernen einer neuen Sprache – in Deinem Fall der Programmiersprache R.
Sieh das ganze als exploratives Ausprobieren einer neuen Umgebung an.
Lass Dich von Fehlermeldungen nicht verunsichern, denn wie schon wie schon Thomas A. Edison, der Erfinder der Glühbirne, sagte:

Voraussetzungen
Um dieses Skript zu verstehen, solltest Du
- R und RStudio installiert haben und
- RStudio mit R verbunden haben und
- Die 4 RStudio-Fenster kennen
Das ist alles in den Digitalen Skripten Installation von R und RStudio und RStudio und R-Skripte beschrieben, schaue gegebenenfalls dort nach und kehre dann hierher zurück!
Am besten öffnest Du parallel zum Digitalen Skript RStudio, so dass Du die im Folgenden vorgestellten Funktionalitäten direkt ausprobieren kannst. Das Digitale Skript kannst Du auch auf einem Smartphone oder Tablet lesen.
Fragen oder Anmerkungen?
Für Anregungen und Kommentare zur Verbesserung dieses Digitalen Skripts ist das RLab-Team immer dankbar! Du kannst auch Fragen zu den Inhalten stellen! Nutze für all das gerne die Kommentar-Funktion!
Vergleich zwischen GUI und IDE
Gerade am Anfang, wenn Du dabei bist R zu lernen, können die skriptbasierte Steuerung und die schier unendlichen Möglichkeiten der statistischen Analysen einschüchternd wirken. Im Vergleich zu anderen Statistiksoftwares wie zum Beispiel Excel oder SPSS verfügt RStudio nicht über ein Graphical User Interface (GUI) sondern über ein Integrated Development Environment (IDE).

Im Vergleich zu dem reinen Programm R, stehen Dir In RStudio das Skriptfenster, Plot-Fenster, Environment und Console zur Verfügung, die Dir die Bedienung erleichtern.
Dennoch ist es eine Voraussetzung, dass Du vorab weißt, was Du berechnen oder darstellen möchtest. In RStudio gibt es nicht die Möglichkeit sich durch die Auswahlmöglichkeiten ,,zu klicken" wie bei SPSS.
Die Steuerung des Programms R ist skriptbasiert und gerade dann ist es wichtig, dass Du Dich nicht von Fehlermeldungen entmutigen lässt. Es ist ganz normal, dass Fehlermeldungen auftauchen oder etwas nicht klappt wie gewünscht.
Allgemeine Informationen zu R-Hilfe-Funktionen findest Du im Digitalen Skript zu R-Hilfe in- und außerhalb von R.
Updates von R, R-Packages und RStudio
Das R Core Team ist verantwortlich für R und stellt ziemlich regelmäßig neue R Versionen online.
Damit Du immer auf dem neusten Stand bist, kannst Du die updateR() Funktion verwenden. Diese ist im R-Package ,,installr" enthalten. Diese Funktion sucht automatisch nach der aktuellsten R Version und bietet dann die Möglichkeit diese zu installieren. So bist du immer auf dem neusten Stand!
Obwohl für die erste Installation von R und RStudio Administratorrechte benötigt werden, sind diese beim Updaten der Programme nicht nötig.
install.packages("installr") # hier wird das Package installiert
library(installr) # hier wird das Package aktiviert
updateR() # Überprüfung auf Aktualität der R Version wird gestartet
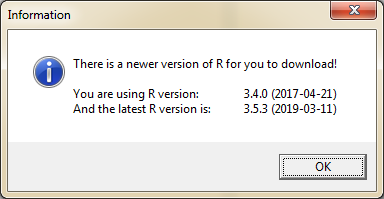
Bist du mit deiner R Version auf dem aktuellsten Stand, wird in der Console FALSE ausgegeben.
Bist bu mit deiner R Version nicht auf dem aktuellsten Stand, erscheint ein neues Fenster. In diesem Fall ist die aktuellste R Version 3.5.3.
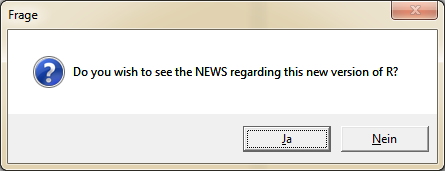
Im nächsten Schritt erscheint ein neues Fenster, in dem Du gefragt wirst, ob Du die ,,News" zu dieser Version dir anschauen möchtest.
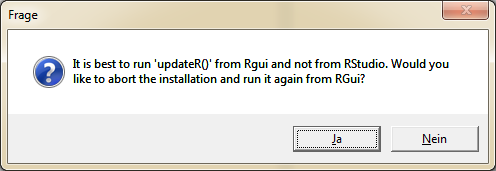
Klickst Du auf ,,Ja", so öffnet sich die Internetseite mit den News zur aktuellsten R Version.
Hier sind die Informationen, bzw. die Erneuerungen der Version zusammengefasst. Diese News findest Du auch bei ,,New features in this version" auf der Download-Seite der jeweils aktuellen R-Version.Um die aktuellste Version von R zu installieren, klicke im darauf folgenden Dialog auf ,,Ja".

Um die neuste R Version zu installieren, ohne RStudio zu verlassen, klickst im nächsten Schritt auf ,,Nein".
Darauf folgt der R Installationsprozess. Es werden hierfür keine Administratorrechte benötigt.
Nachdem der Installationsprozess abgeschlossen ist, erscheint ein neues Fenster, in dem gefragt wird, ob die Packages von der alten R Version in die neue R Version kopiert werden sollen.
Nun hast Du 2 Möglichkeiten: Entweder folgst du dem nächsten Dialogfenster oder Du kopierst die Packages ,,per Hand" (siehe unten).
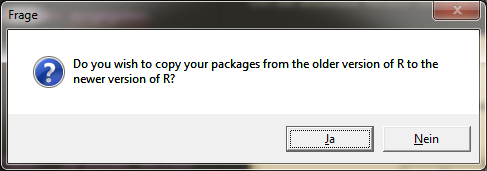
Hier kannst Du entscheiden, ob die Packages in der alten R-Version erhalten bleiben sollen oder nicht.
Achtung! Der Kopiervorgang kann ein Weilchen dauern, je nachdem wieviele Packages in der R Library enthalten sind.
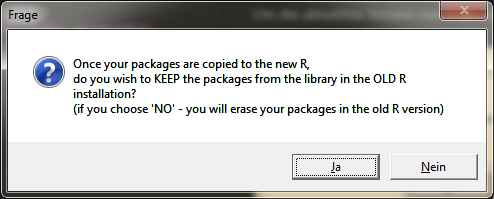
Ist der Kopiervorgang oder der Verschiebevorgang beendet, erscheint die Frage nach dem Update der Packages.
Du findest den library Ordner dort, wo deine R Version gespeichert ist. 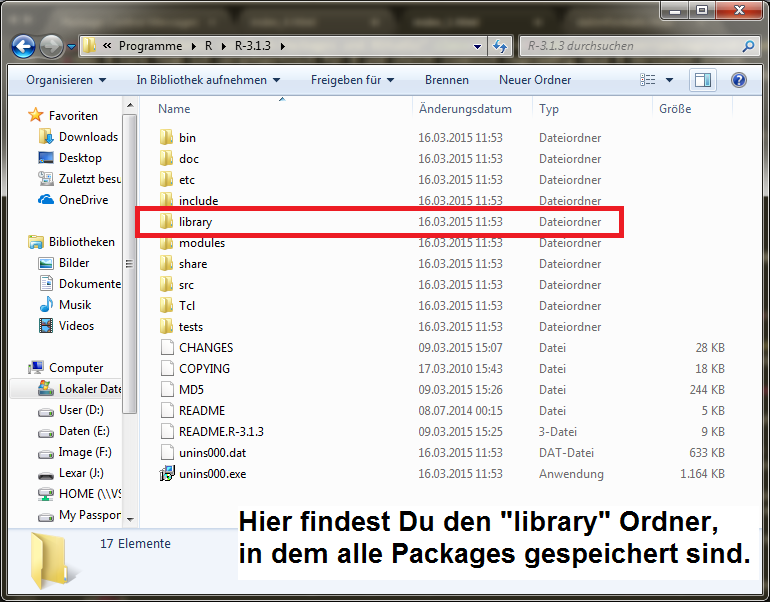
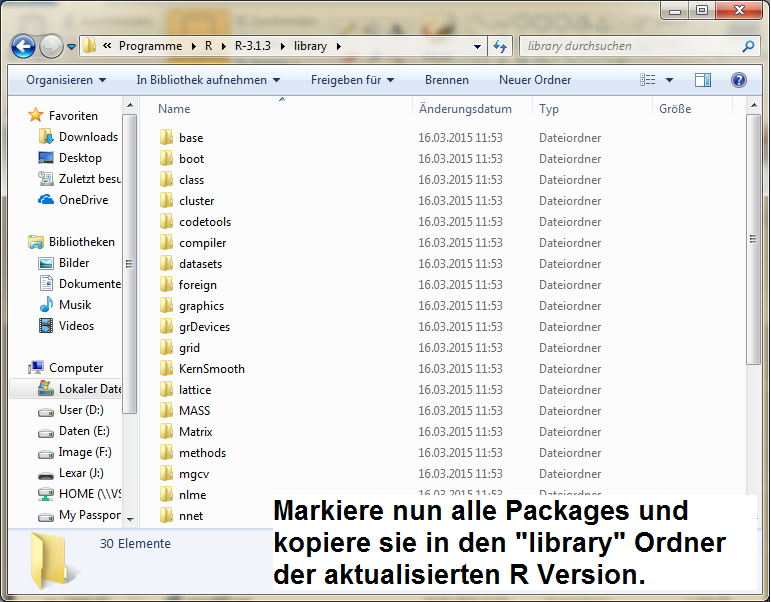
Ist dieser Vorgang abgeschlossen, musst Du in den Global Options die ,,neue" R Version auswählen. Anschließend RStudio einmal schließen und dann erneut öffnen.
Fertig! Du bist mit Deiner R Version und deinen R-Packages wieder auf dem neusten Stand!
Um Fehlermeldungen zu vermeiden, empfielt es sich die ,,alte" R Version vom Computer/Stick zu löschen.
Du kannst R auch relativ einfach ,,manuell" updaten: Dazu installierst Du einfach die neueste Version (z.B. in ein Verzeichnis ,,R_Version_X") und kopierst dann Deine Packages wie oben im Tab ,,Packages per Hand kopieren" beschrieben. Nachteile: Du musst mehr mitdenken und kannst Dich nicht bequem durch den Update-Prozess klicken. Vorteile: Du sparst das installr-Package, hast die volle Kontrolle und verstehst besser, wie die R-Installation strukturiert ist.
Du möchtest RStudio aktualisieren?
Mit der Funktion install.RStudio() wird deine RStudio Version aktualisiert. Diese Funktion findest Du auch im Packages ,,installr".
Hinweis: Die Funktion prüft nicht die Aktualität deiner Version, sondern überschreibt einfach die vorhandene Version.
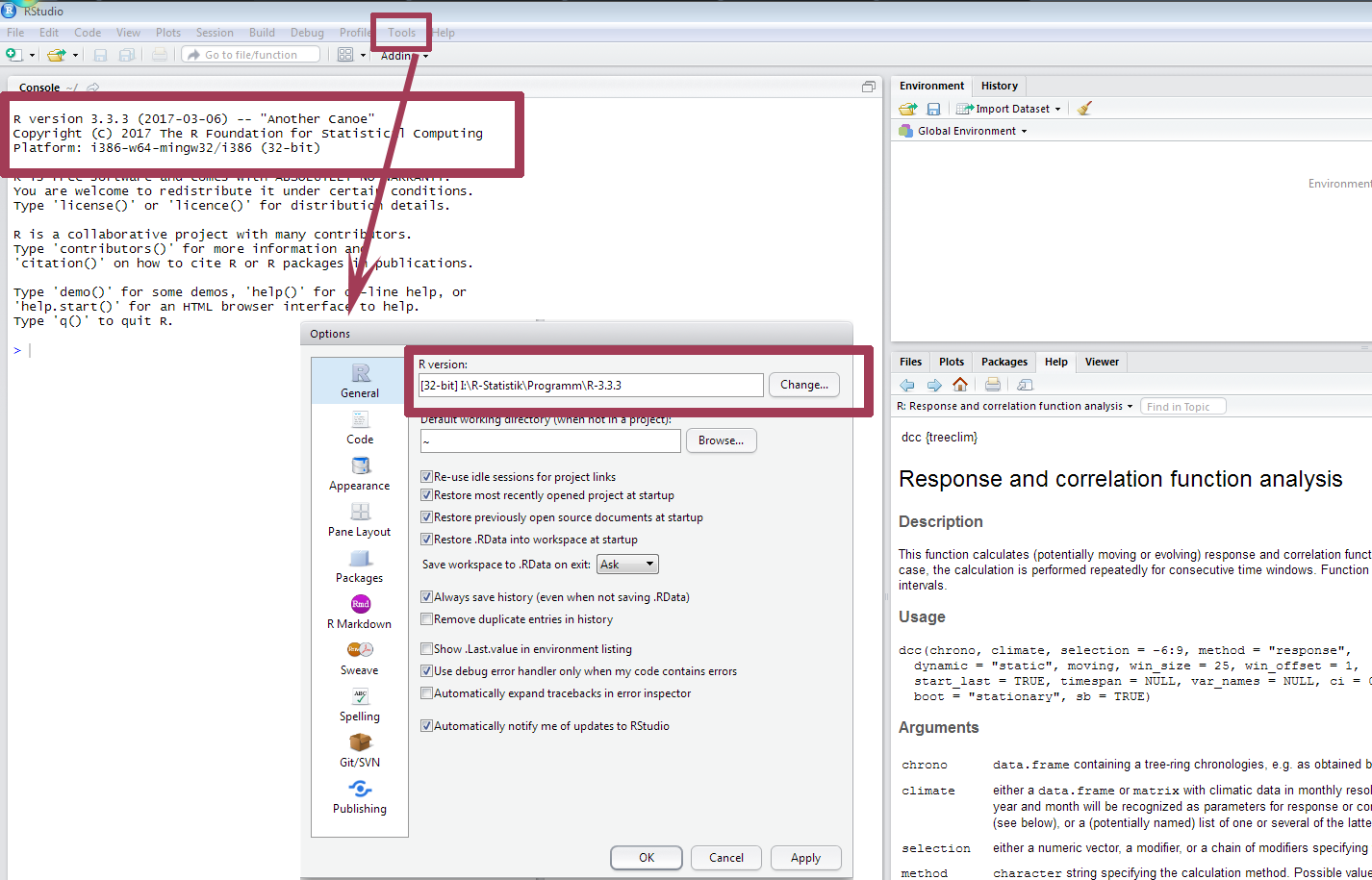
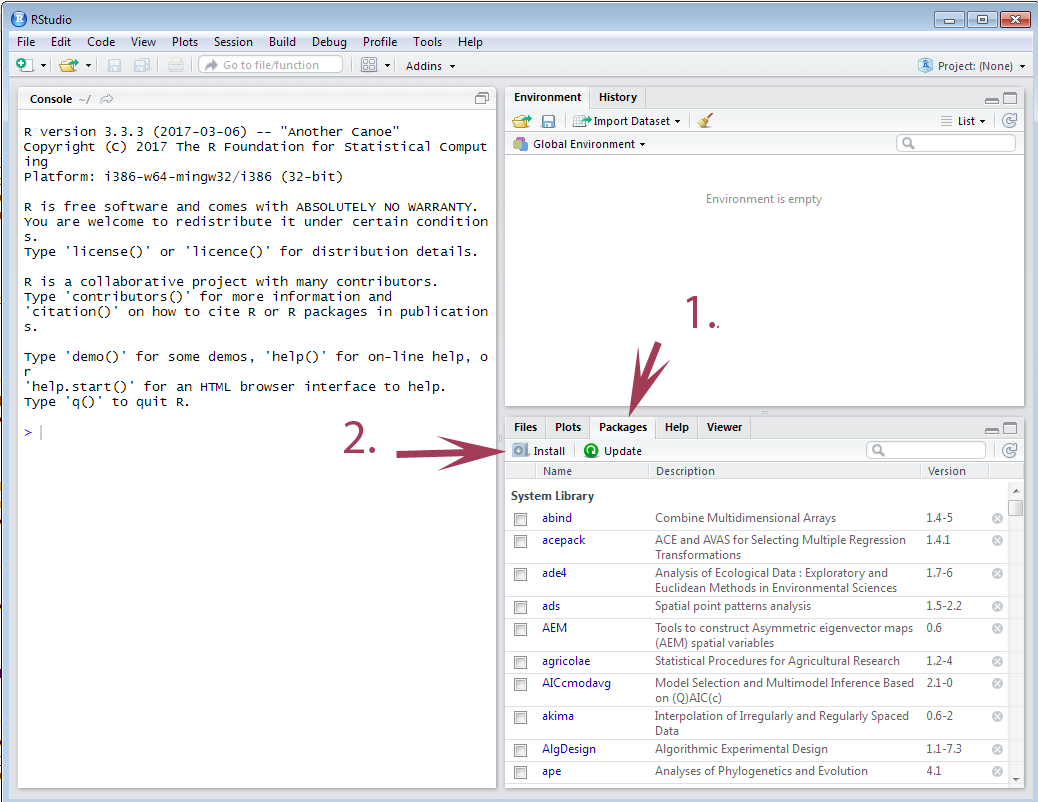
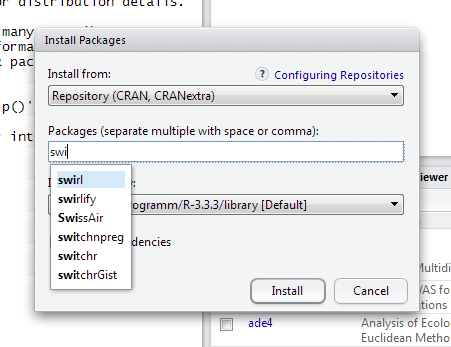
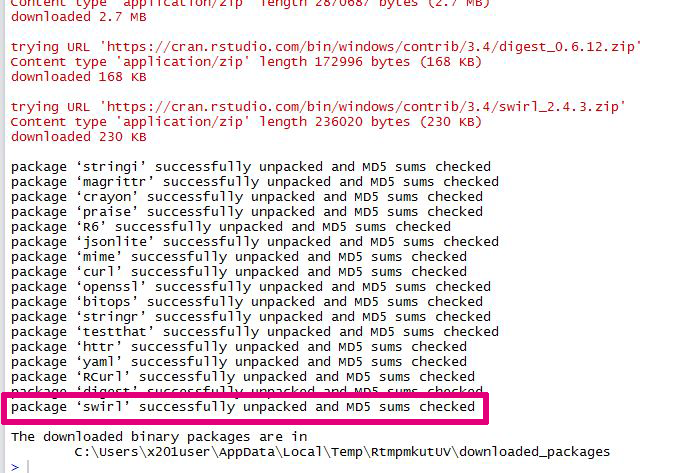
Installation eines R-Packages
In dieser Slideshow kannst Du Dir anschauen, wie schnell und einfach Du ein R-Package zu R hinzufügen kannst!
Alternative Package-Ressourcen und Installation:
Es gibt R-Packages auch als Zusatzmaterial zu R-Büchern oder Fachartikeln, die oft auf Portalen wie R-Forge oder GitHub veröffentlicht werden. Unter anderem da die Regeln zur Veröffentlichung auf CRAN (The Comprehensive R Archive Network) recht streng sind, werden nicht alle Packages dort publiziert.
Für die Installtion von R-Forge kanninstall.packages()so verwendt werden:install.packages("spdyn", repos="http://R-Forge.R-project.org")# "spdyn" = Beispiel-Packagename
Die Installtion von GitHub geht mit folgendem Code:
library(devtools) # ggf. zuvor einmalig install.packages("devtools")
install_github("andrewzm/STRbook") # "GitHub-Account-Name/Package-Name"
Auch für Packages von Bioconductor existiert eine eigene Installationsroutine.
R-Packages werden zum Teil (auch auf CRAN) als .zip-Dateien bereitgestellt und können zum Beispiel mit folgendem Code installiert werden:
install.packages('D:/ORDNER/PACKAGE.zip', repos = NULL)
Alternativ kannst Du die .zip-Datei auch selbst entpacken und den entpackten Ordner in den library-Ordner Deiner R-Installation kopieren.
Generell kann die Verwendung voninstall.packages()Vor- und Nachteile haben, abhängig von Deiner Arbeitsweise, dem Zweck Deines Codes usw. Zumindest sollte der Code im Skript auskommentiert werden, um zu vermeiden, dass das Package jedes Mal neu installiert wird, wenn Du den Code ausführst.
Hintergrundinfos zu R-Packages:
- Álvarez, A. / DataCamp (2017) R Packages: A Beginner's Guide [26.03.2019]
- CRAN Contributed Packages [26.03.2019]
- Grolemund, G. / RStudio Quick list of useful R packages [26.03.2019]
- STHDA Installing and Using R Packages [28.06.2019]
- Wickham, H. (2018) R packages book [26.03.2019]
Fragen oder Anmerkungen?
Für Anregungen und Kommentare zur Verbesserung dieses Digitalen Skripts ist das RLab-Team immer dankbar! Du kannst auch Fragen zu den Inhalten stellen! Nutze für all das gerne die Kommentar-Funktion!
Abspeichern von Ergebnissen
Wie können Ergebnisse einer Analyse oder einer Berechnung abgespeichert werden, um zu einem späteren Zeitpunkt weiter mit ihnen zu arbeiten?
Wie können Ergebnisse aus R herausgeschrieben werden, um sie in einem anderen Programm weiter bearbeiten zu können?
Im Folgenden geht es um die Speicher-, bzw. Lade-Optionen von:
- allen Objekten im Environment
- Consolen-Output
Objekte aus dem Environment abspeichern
Bestimmt ist Dir beim Schließen von R schon einmal aufgefallen, dass Du gefragt wirst, ob du den ,,Workspace" sichern möchtest. Diese Datei hat die Endung .RData.
Folgende Abbildung wird Dir bekannt vorkommen.
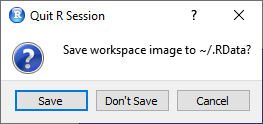
Wenn Du hier auf ,,save" klickst, wird Dein gesamter Workspace dort abgespeichert, wo Deine Working Directory gesetzt wurde. In diesem Fall wurde die Working Directory nicht gesetzt, sodass der Workspace auf dem Computer im Verzeichnis ,,Dokumente" gespeichert wurde.
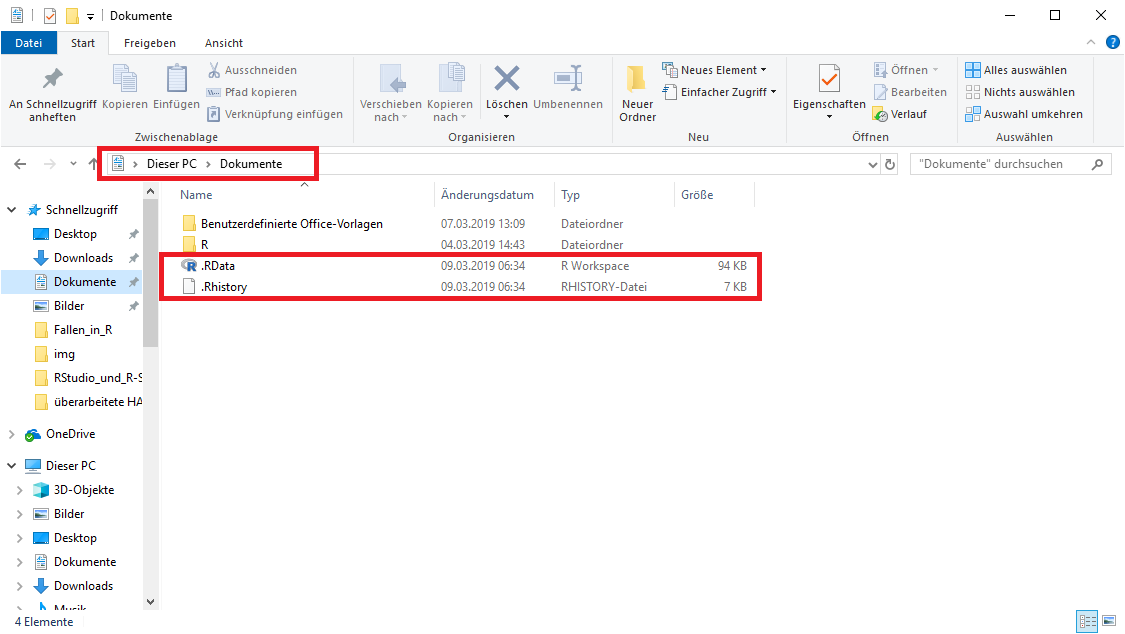
Dieses Vorgehen hat verschiedene Nachteile:
- Beim Speichern ohne ,,richtigen" Dateinamen wirst Du den gespeicherten Inhalt dieser Datei nur schwer wieder zum dazugehörigen Skript zuordnen können.
- Beim erneuten Öffnen von R und RStudio wird die Datei an diesem Speicherort ,,automatisch" wieder in das Environment geladen. Der Grund hierfür liegt daran, dass alle Dateien aus diesem Verzeichnis geladen werden. Hier sind auch die 'Packages' gespeichert.
- Beim erneuten Abspeichern eines Workspaces wird die vorherige Datei überschrieben, ohne dass es eine Warning-Message gibt.
- Arbeitest Du an einem Computer in der Uni, wirst Du Deine Datei nicht wiederfinden können, ist der Computer einmal runtergefahren worden. Auf den Computern in der Uni werden alle Dateien, die nicht im Kursverzeichnis gespeichert worden sind, beim Runterfahren gelöscht.
Best practice
Um den gesamtes Environment, also den Workspace (interner Speicher von R) zu speichern, wird die Funktion save.image() verwendet.
Hier kannst Du den Speicherpfad bestimmten und auch einen Dateinamen vergeben
save.image("F:/R/Ergebnisse_Korrelation/Korrelation.RData").
Es ist empfehlenswert diesen ,,Code-Schnipsel" an das Ende eines jeden Skriptes zu kopieren. So hast Du immer die Möglichkeit deinen Workspace zu speichern.
Das Setzen einer Working directory kann Ergebnisse retten. So wird der Workspace beim Schließen wenigstens im entsprechenden Verzeichnnis gespeichert.
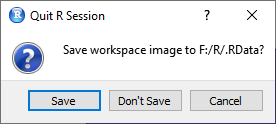
Objekte in das Environment laden
Um die den Workspace wieder in R zu laden wird die Funktion load() verwendet.
load("F:/R/Ergebnisse_Korrelation/Korrelation.RData")Du öffnest RStudio und es stehen bereits Dateien im Environment?
Es wurde ein Workspace (.RData) geladen.
Mit der Funktion rm(), (engl. remove = entfernen), kannst Du alle Dateien auf einmal im Environment löschen.
rm(list = ls()) # löscht alles im Environment/WorkspacesDiese Aktion lässt sich nicht rückgängig machen.
Auch hier ist es empfehlenswert, diesenConsolen-Output abspeichern
Um die Struktur eines Objektes zu speichern, kann man den gesamten Consolen-Output mit der Funktion 'sink()' in eine Datei schreiben.
Diese kann dann zum Bepiel mit dem Editor geöffnet und weiterbearbeitet werden.
Die Funktion sink() leitet den Output der Console in eine Datei um. Die Ausgabe erfolgt während der aktiven Umleitung nur in die Ausgabe-Datei und nicht mehr in die Console. mit dem erneuten Befehl sink() wird die Umleitung deaktiviert und die Ausgabe erfolgt wieder in der Console.
sink("F:/R/Ergebnisse_Korrelation/Korrelation.txt") # Umleitung wird aktiviert
# Dateiname und Dateiendung werden festgelegt
cor(sat.act[1:200,2:3] ) # Korrelationsergebnis würde eigentlich in der Console ausgegeben werden
sink() # Umleitung wird deaktiviert
file.show("Korrelation.txt") # Datei wird in einem neuen Fenster angezeigt.Abspeichern von Plots
Genauso wie Tabellen und andere Ergebnisse, müssen auch Grafiken (Plots)
Im folgenden Abschnitt werden Dir 2 Möglichkeiten vorgestellt, wie Du erstellte Grafiken aus RStudio exportieren kannst.
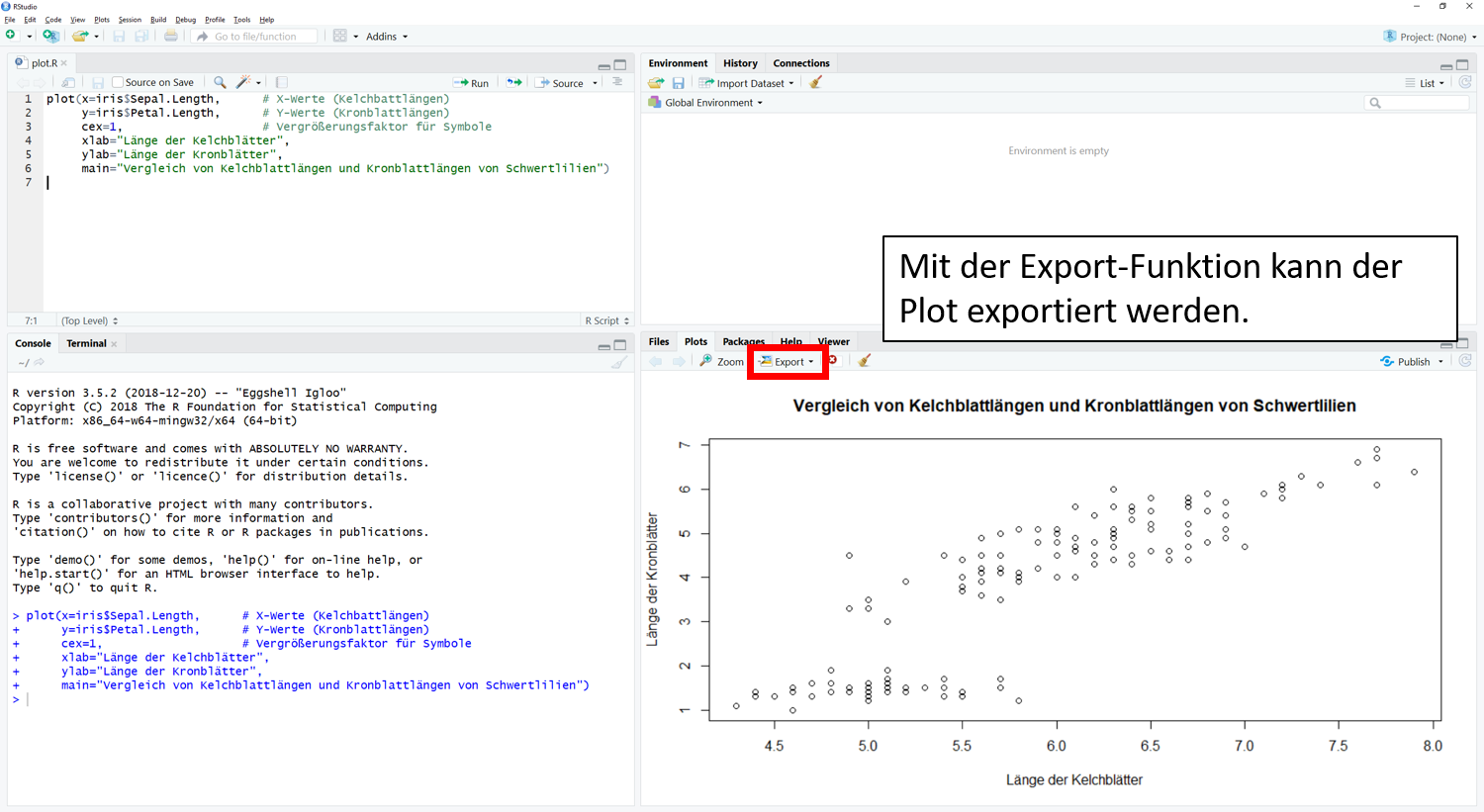
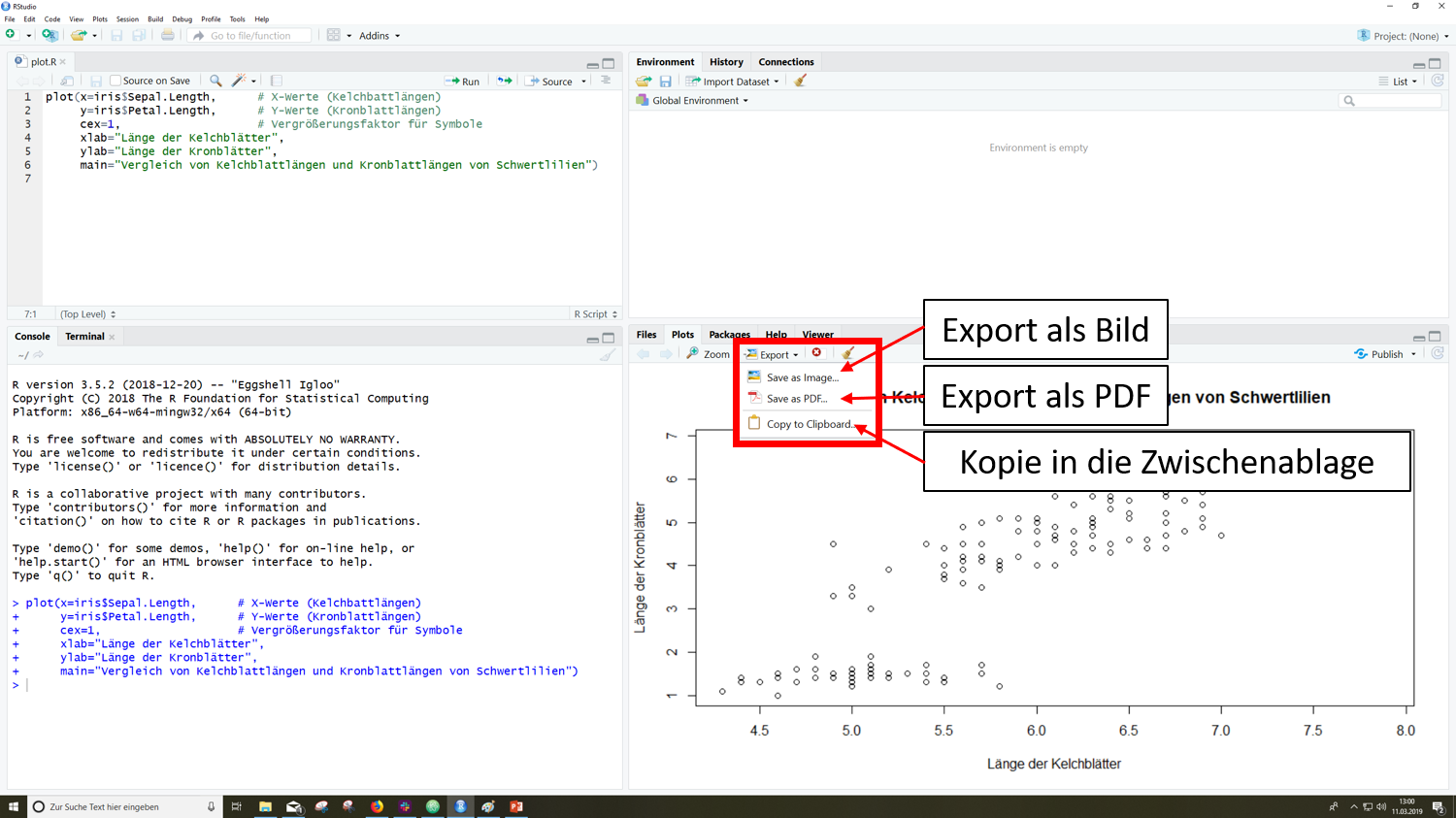
Abspeichern über das IDE (Integrated Development Environment)
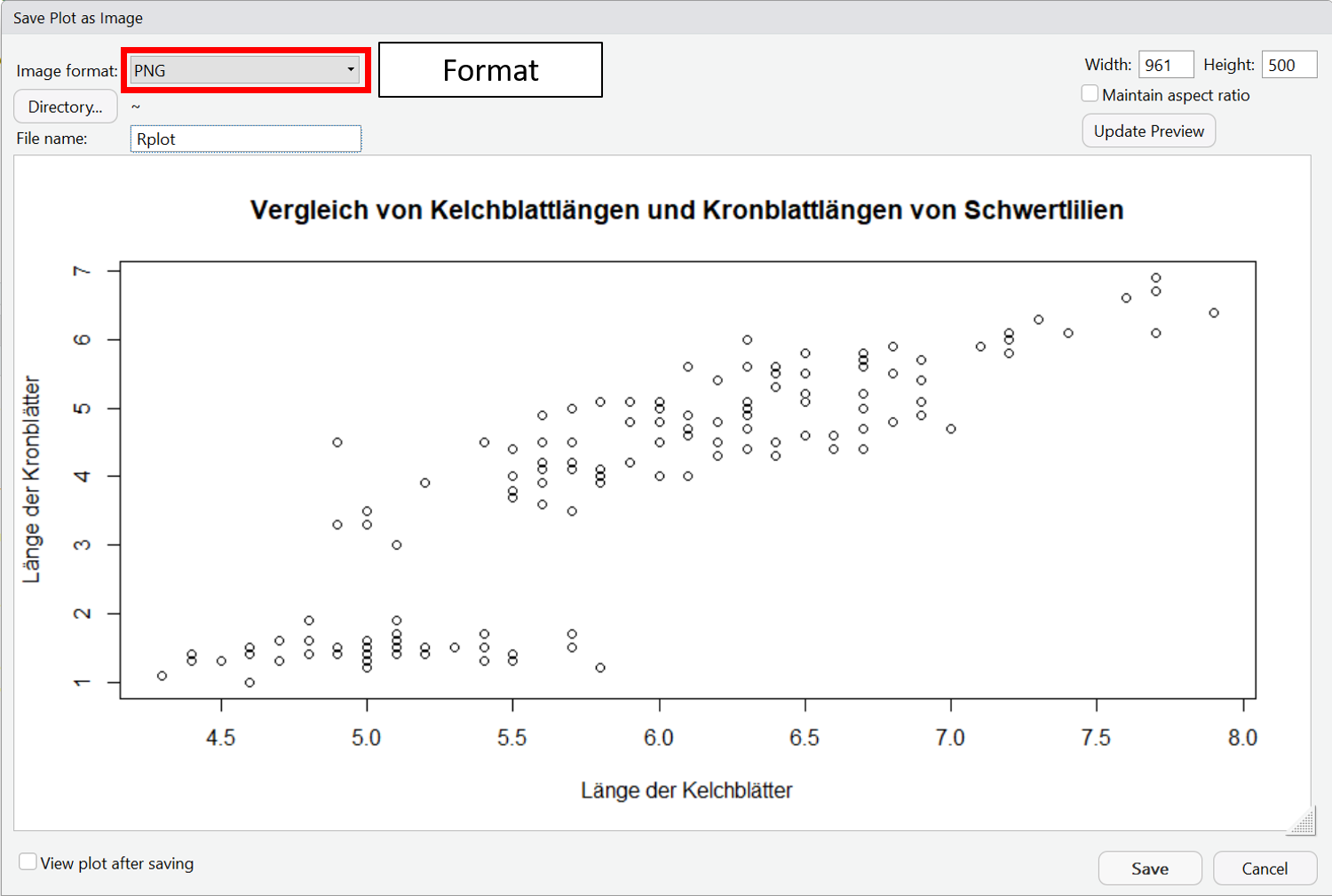
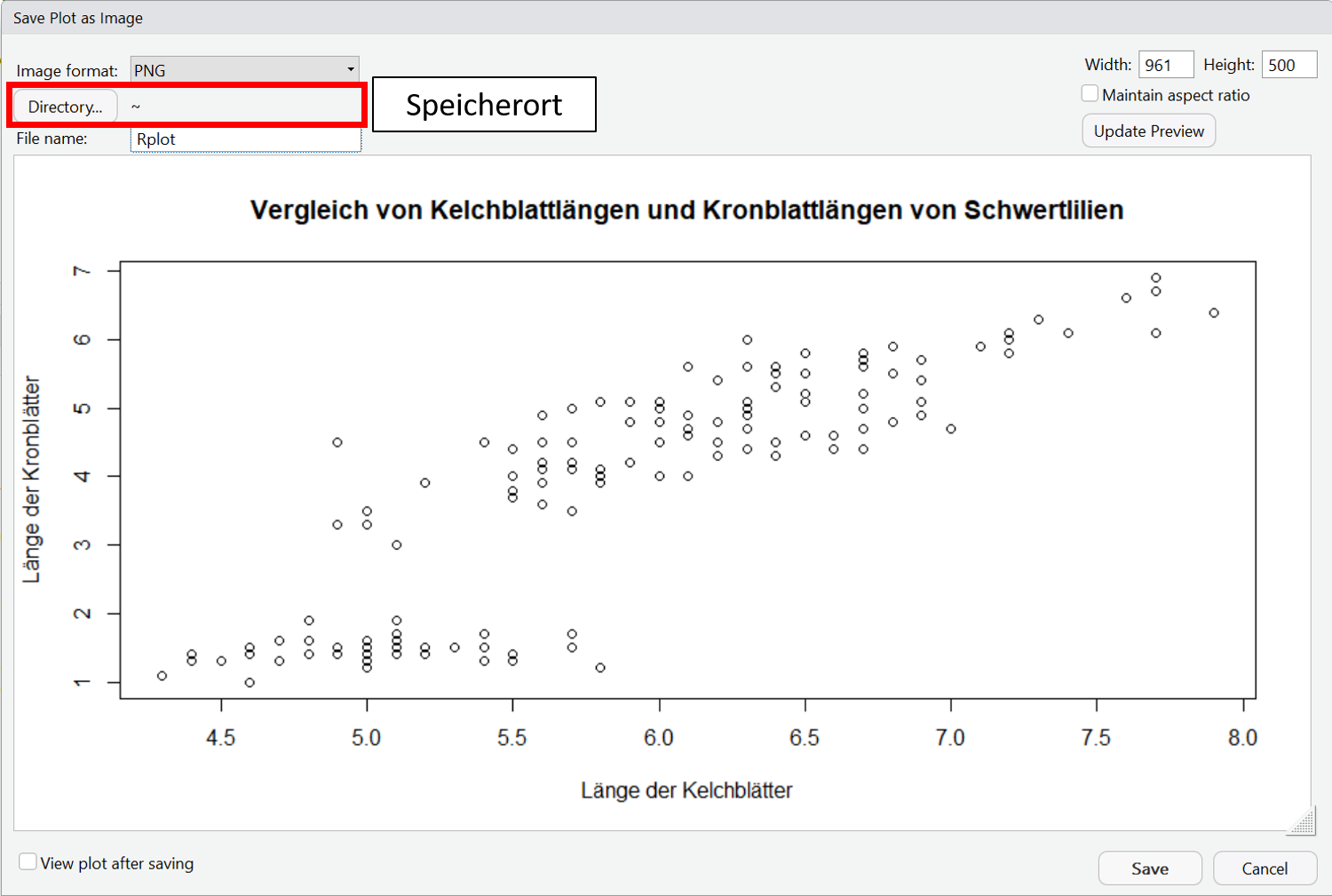
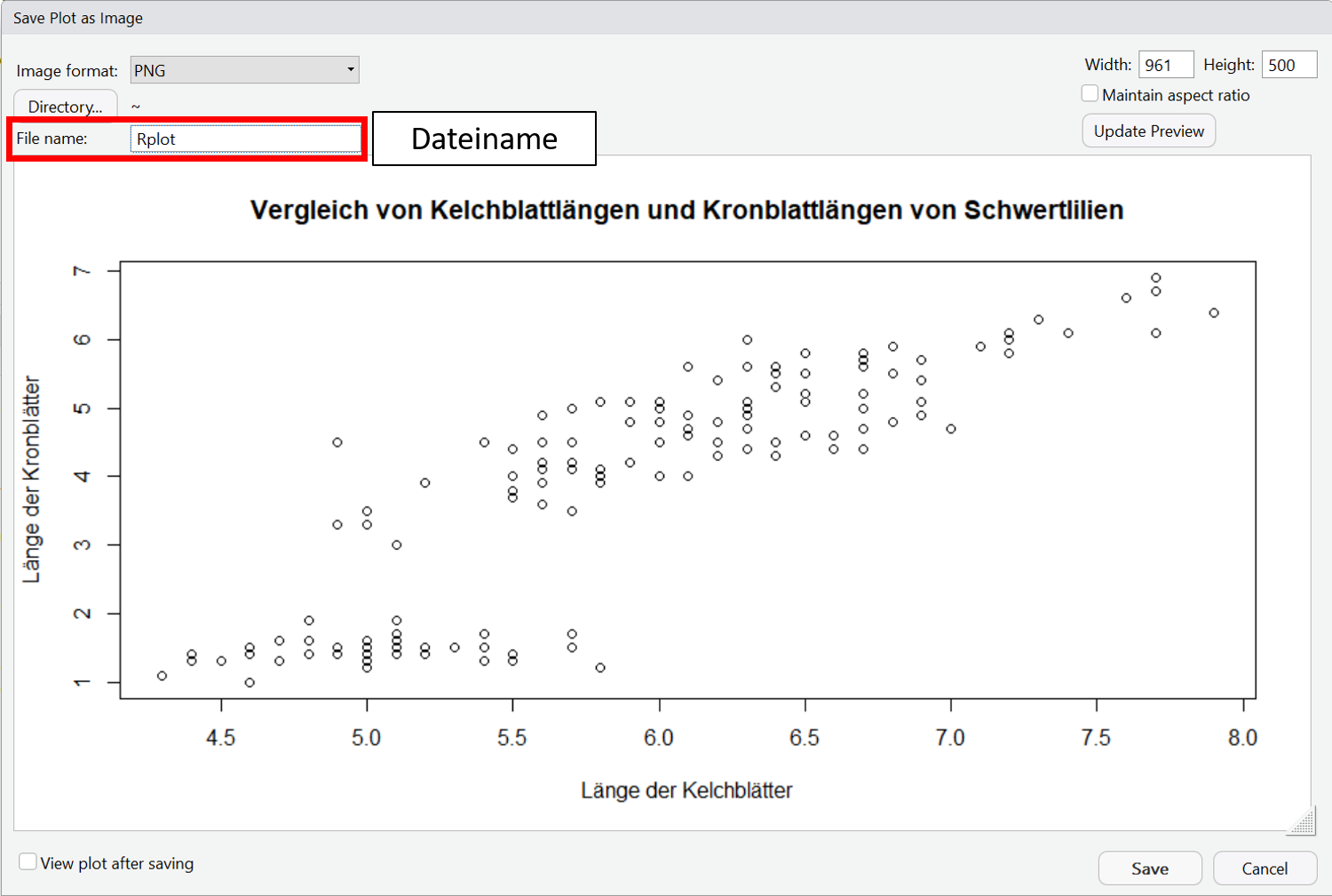
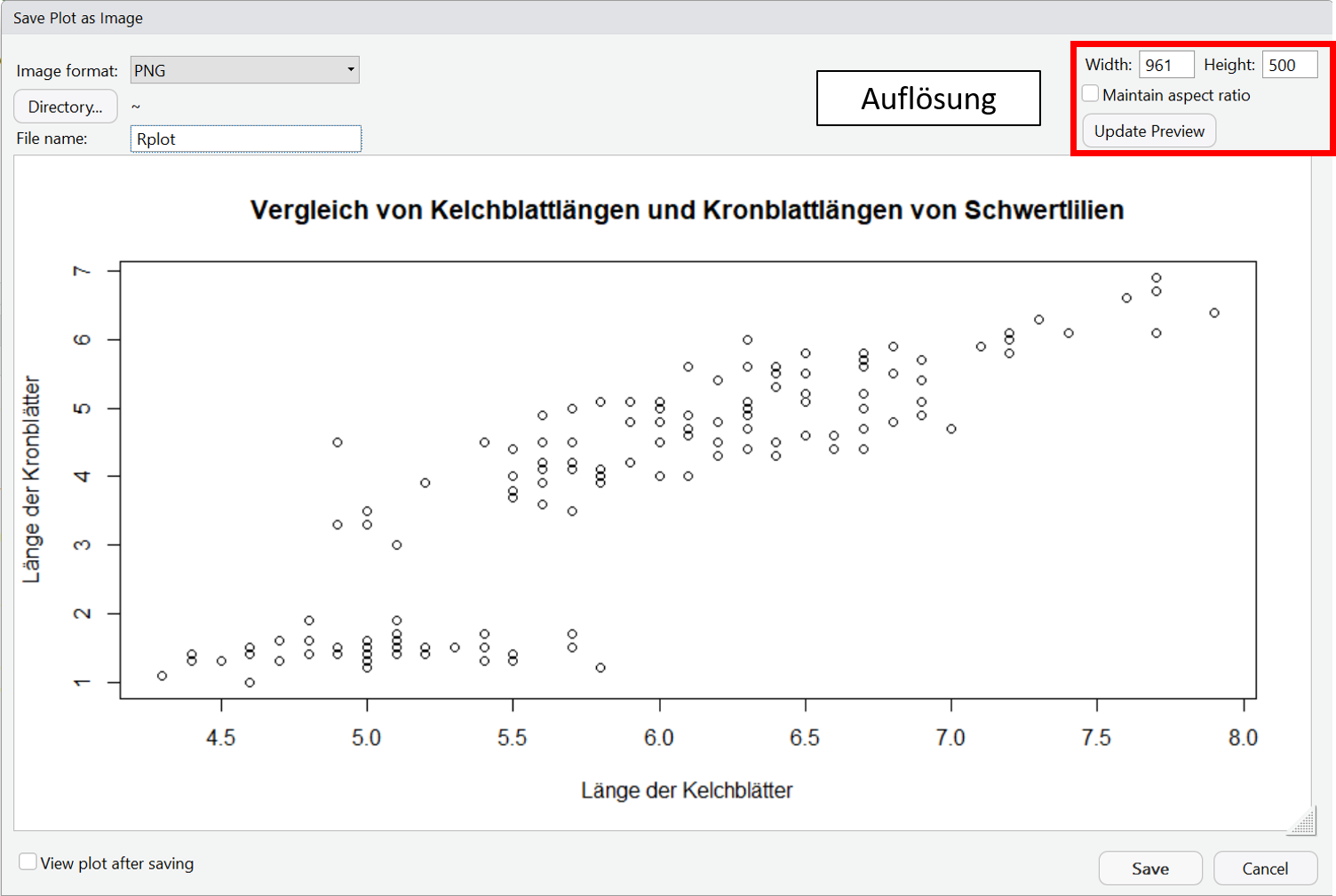
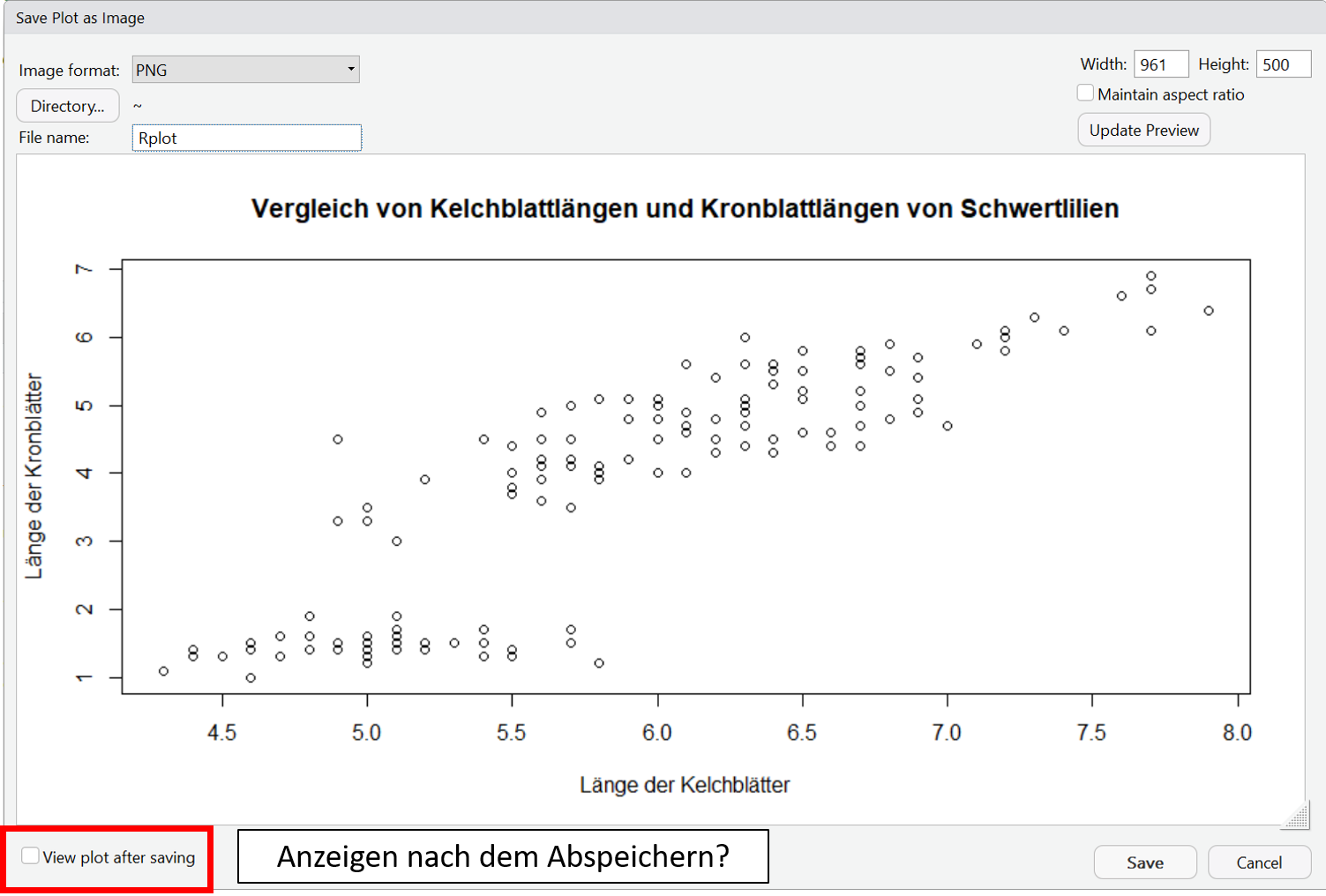
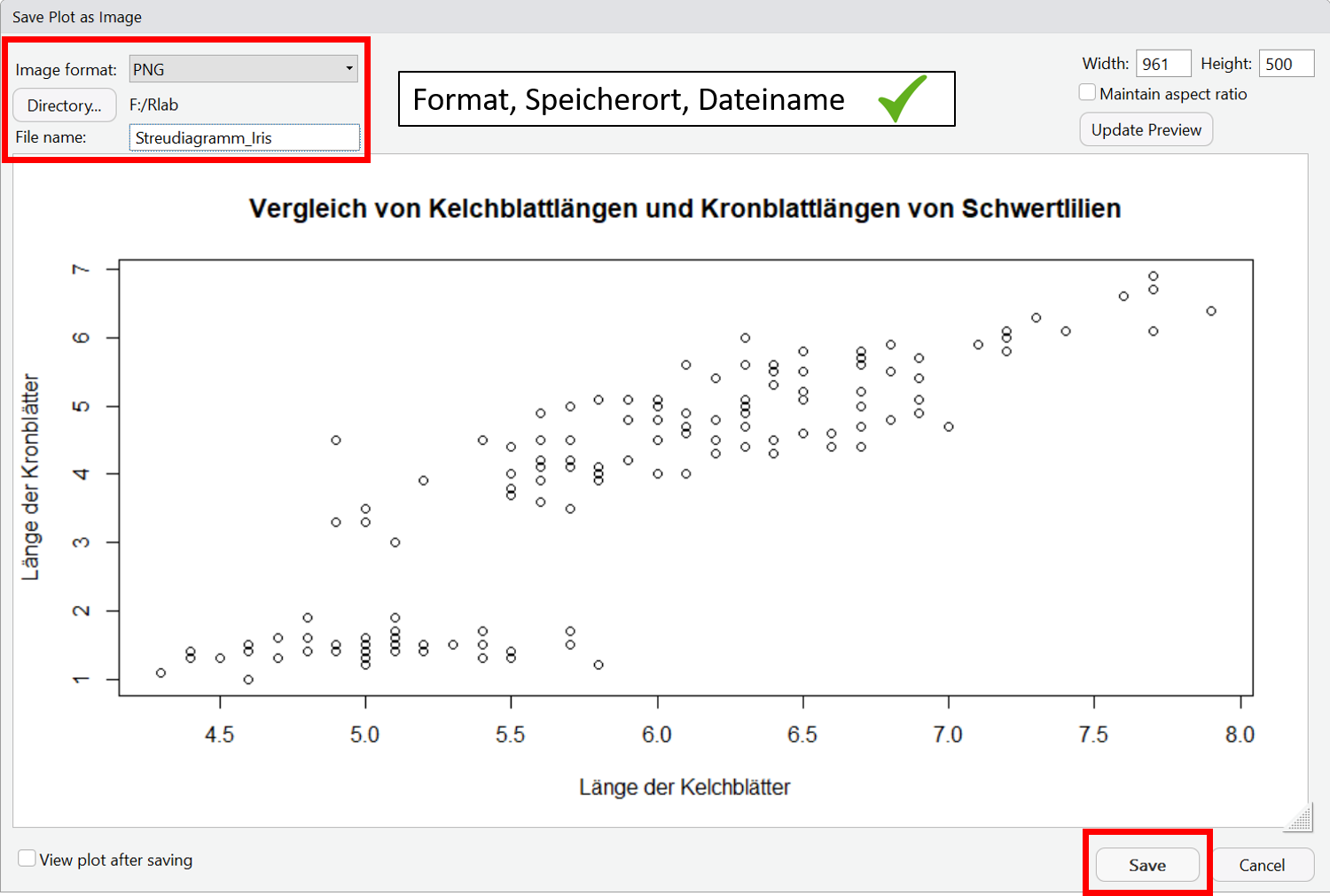
In RStudio gibt es die Möglichkeit erstellte Plots über das Plot-Fenster über den Menüpunkt ,,Export" zu exportieren. Hier kannst Du zwischen verschiedenen Dateiformaten wählen.
Eine Schritt-für-Schritt Anleitung siehst Du in der folgenden Slideshow:
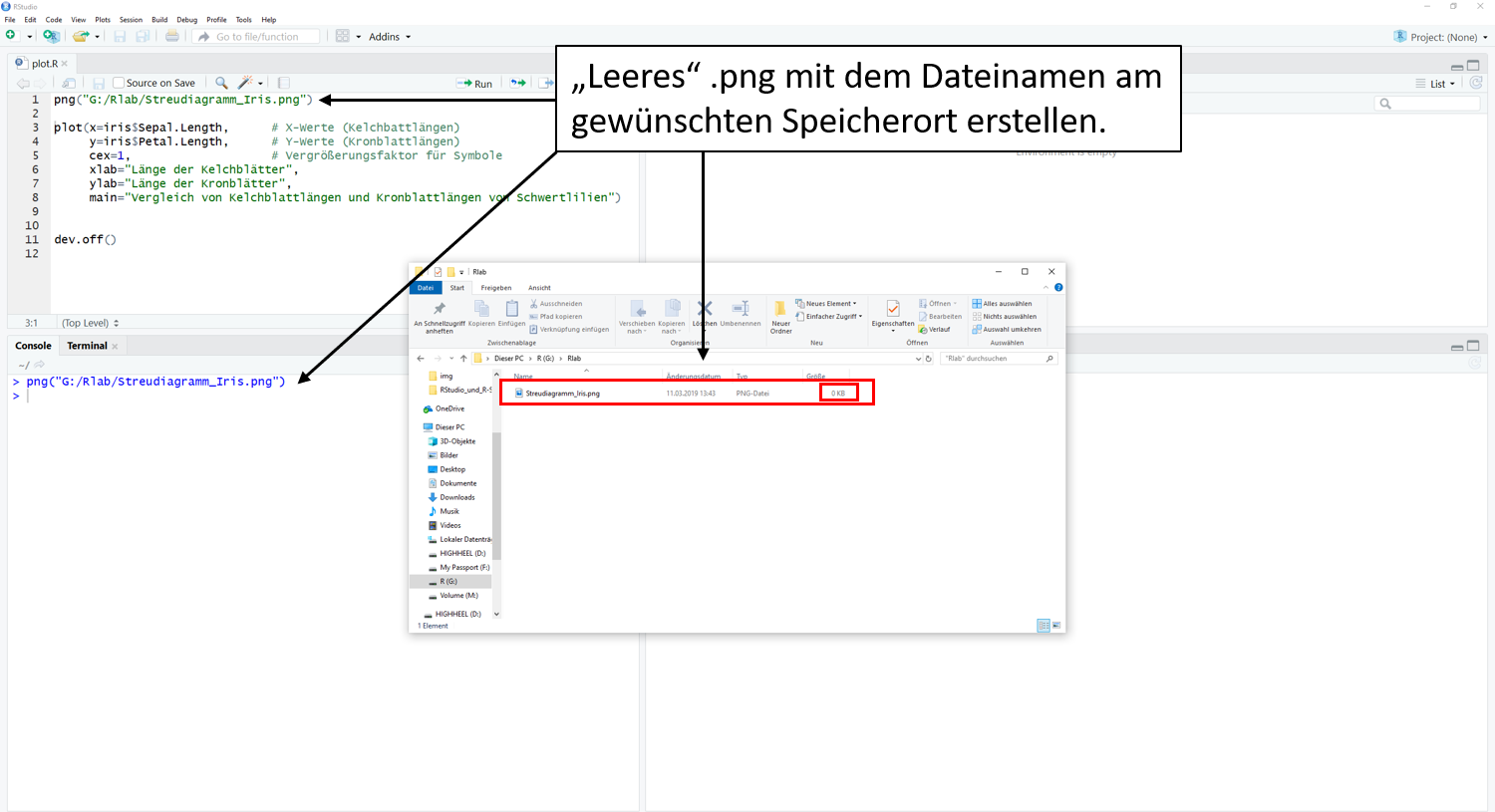
Abspeichern mit der Funktion png()
Desweiteren hast Du die Möglichkeit den Export auch zu skripten.
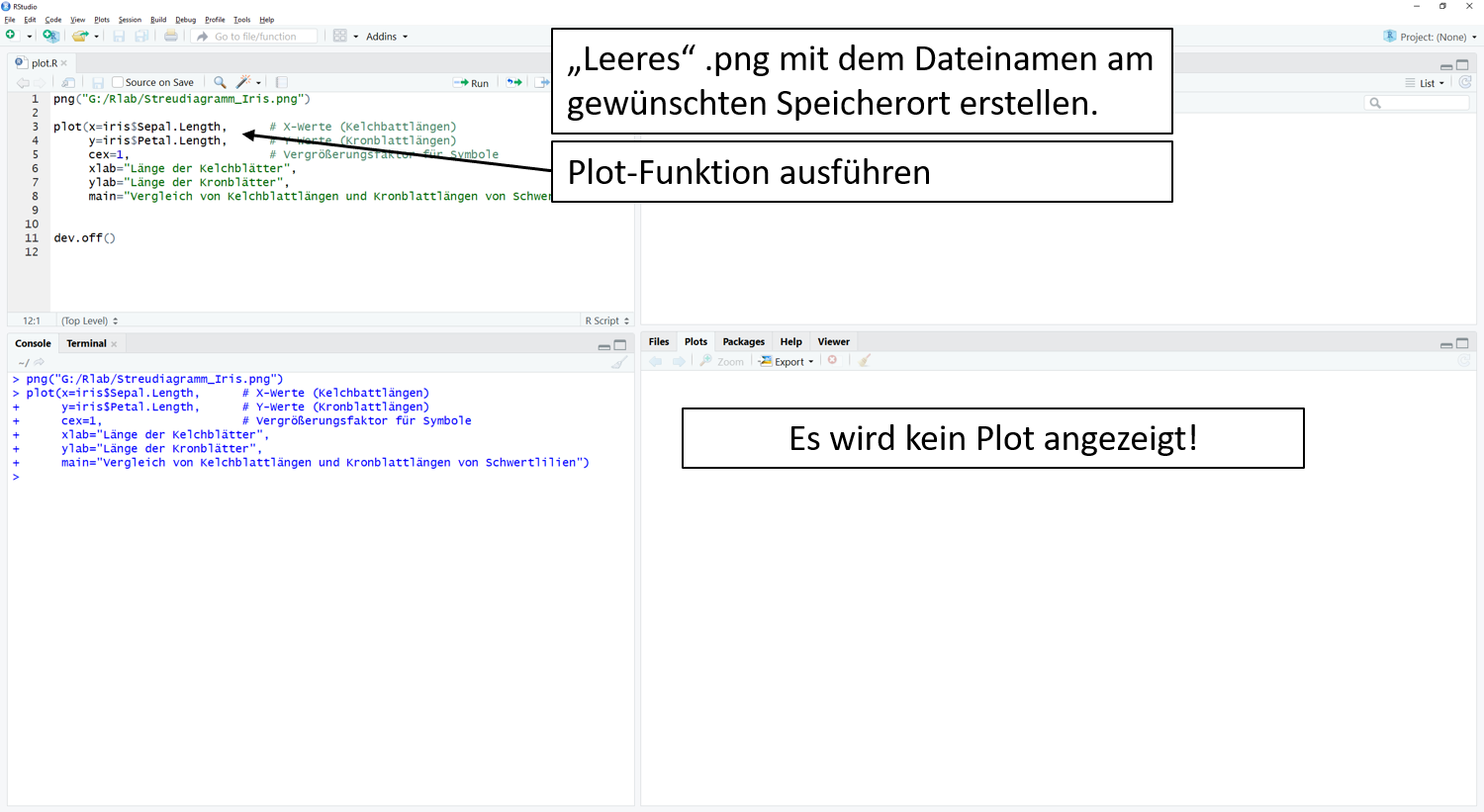
Im ersten Schritt wird eine leere Datei (hier .png) erstellt.
Im zweiten Schritt folgt die eigentliche plot() Funktion. Hierbei wird der Plot aber nicht im Plotfenster dargestellt. R behält den Plot im unsichtbaren Zwischenspeicher.
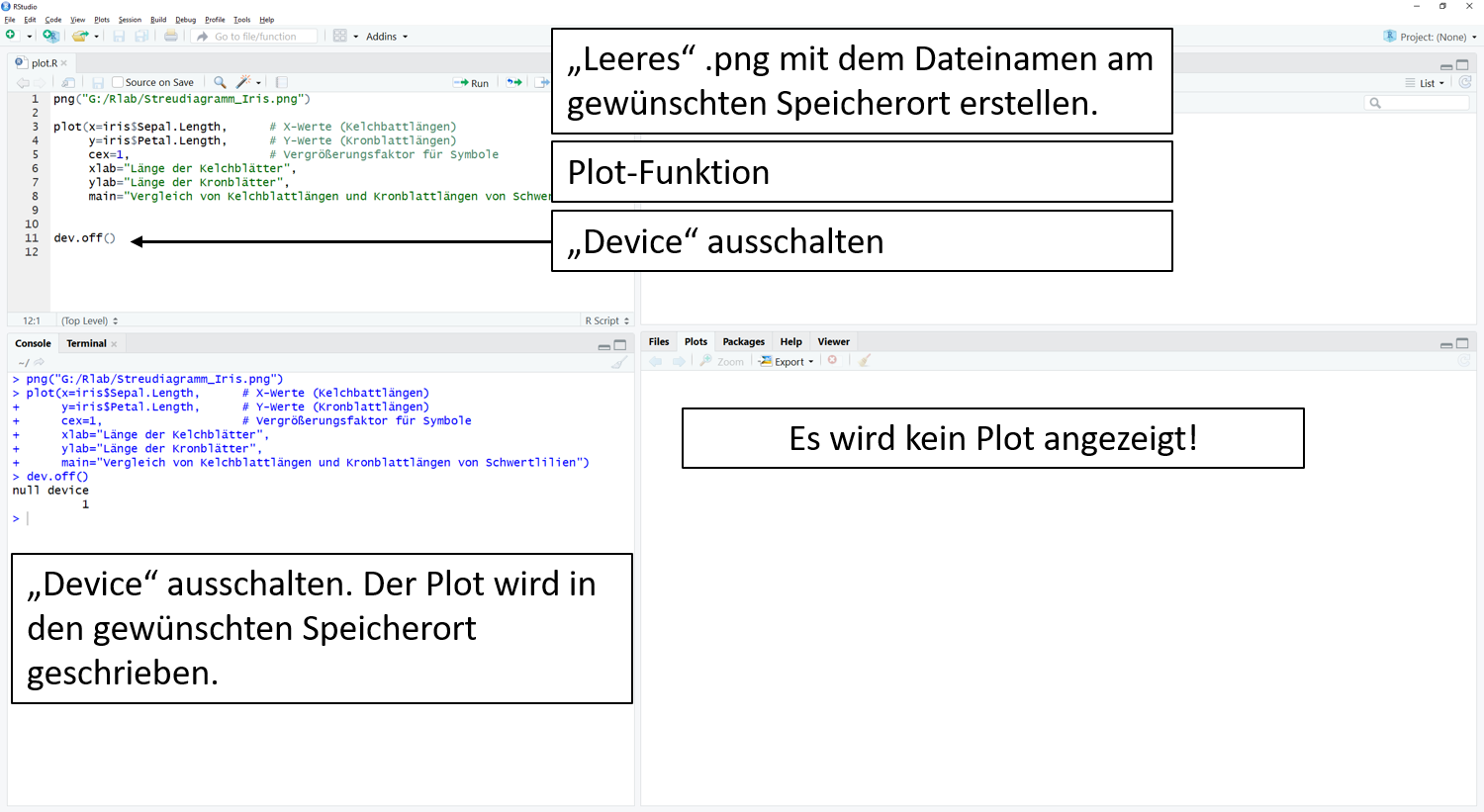
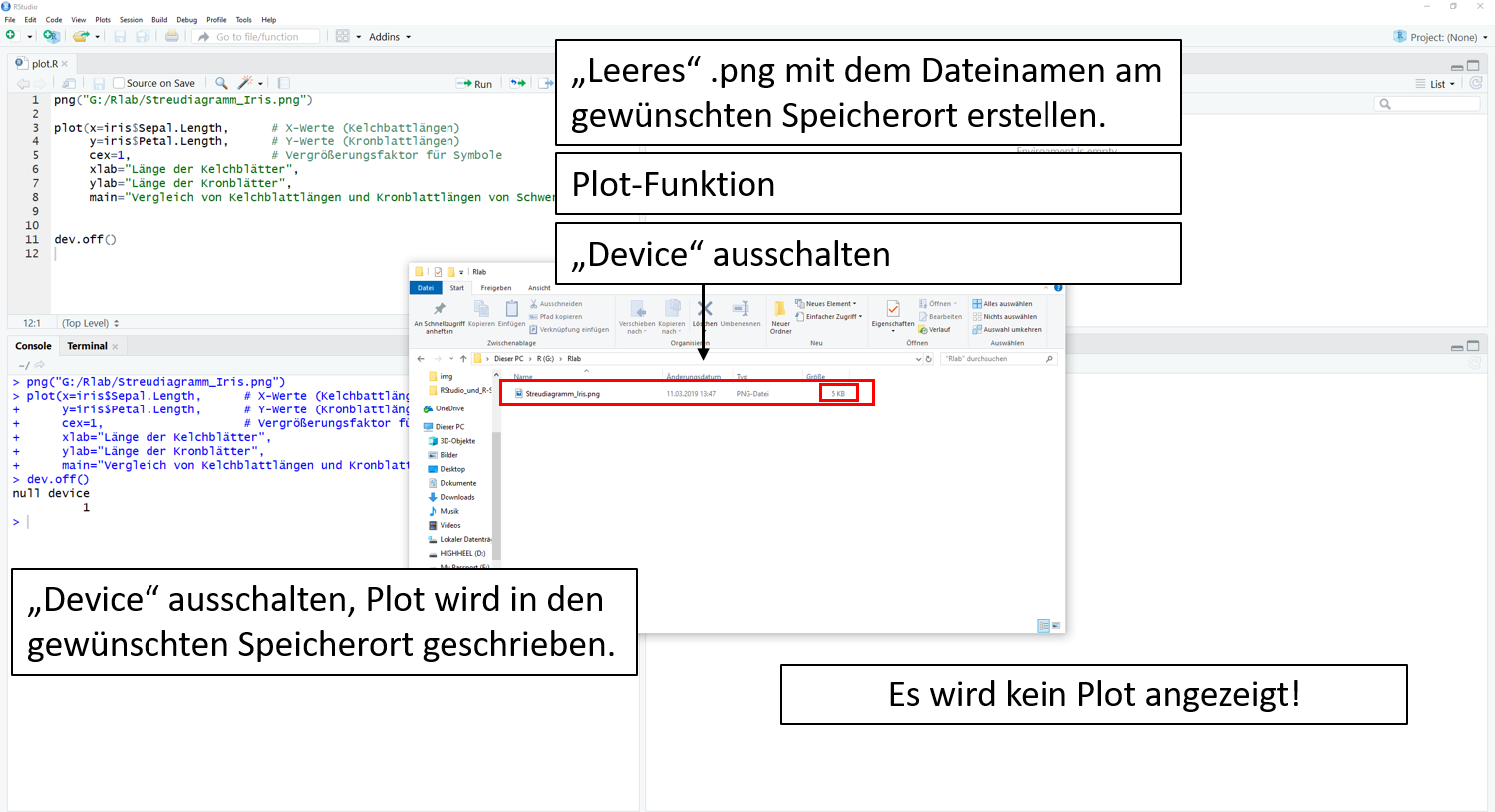
Im dritten Schritt wird der Inhalt des unsichtbaren Zwischenspeichers mit dem Befehl dev.off() in die leere Datei aus Schritt 1 geschoben.
Vorsicht: Wird der Befehl wiederholt oder ein anderer plot() Befehl ausgeführt, wird die Datei jedes Mal wieder überschrieben. Es wird immer der letzte plot() Befehlsinhalt vor dem dev.off()-Befehl in die Datei geschoben.
Du möchtest den Export gleich in R ausprobieren?
In der Box findest Du unter dem Reiter Code den Code (den Du Dir so auch gleich nach R kopieren kannst) und unter dem Reiter Console, findest du den Consolen-Output.
# Leere png Datei wird erstellt.
png("G:/Rlab/Streudiagramm_Iris.png", width = 10, height = 10, units="cm", res = 600) # Hier werden Größe und Auflösung der Gafik festgelegt
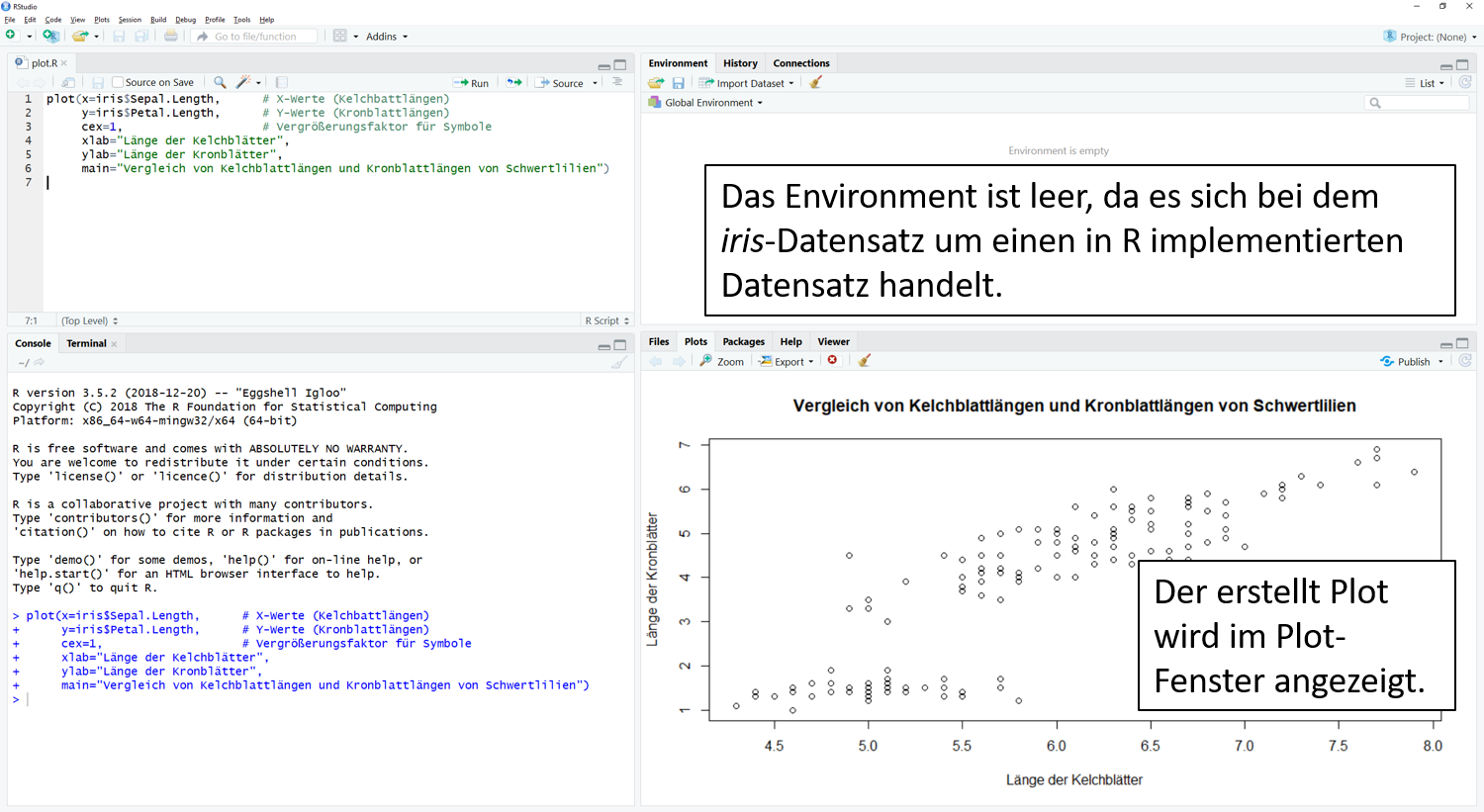
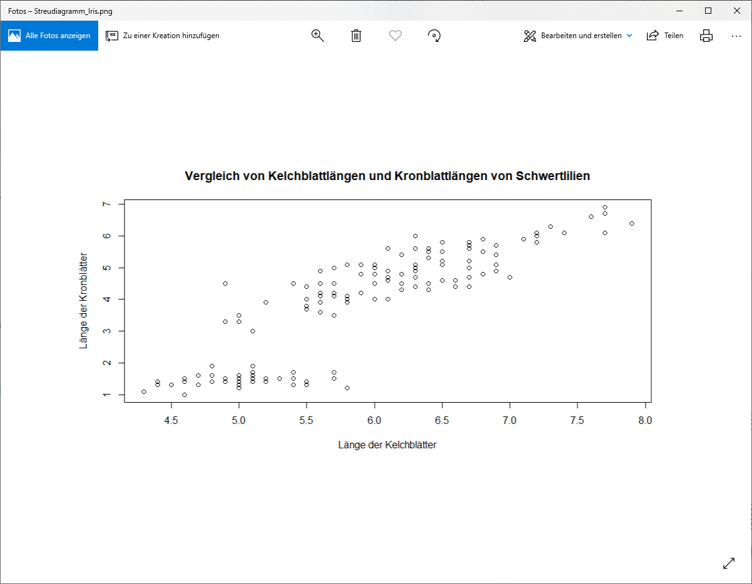
plot(x = iris$Sepal.Length, # X-Werte (Kelchbattlängen)
y= iris$Petal.Length, # Y-Werte (Kronblattlängen)
cex = 1, # Vergrößerungsfaktor für Symbole
xlab = "Länge der Kelchblätter", # X-Achsenbeschriftung
ylab = "Länge der Kronblätter", # Y-Achsenbeschriftung
main = "Vergleich von Kelchblattlängen und Kronblattlängen von Schwertlilien") # Titel der Abbildung
dev.off()
# Leere png Datei wird erstellt.
> png("L:/Rlab/Streudiagramm_Iris.png")
>
> plot(x = iris$Sepal.Length, # X-Werte (Kelchbattlängen)
+ y = iris$Petal.Length, # Y-Werte (Kronblattlängen)
+ cex = 1, # Vergrößerungsfaktor für Symbole
+ xlab = "Länge der Kelchblätter",
+ ylab = "Länge der Kronblätter",
+ main = "Vergleich von Kelchblattlängen und Kronblattlängen von Schwertlilien")
>
> dev.off()
null device
1
>
Eine Schritt-für-Schritt Anleitung siehst Du in der folgenden Slideshow:
Abspeichern von Grafiken in anderen Dateiformaten:
Weitere nützliche Hinweise zum Abspeichern von Grafiken in anderen Dateiformaten (.pdf und .jpeg) findest Du auf Seite 539f im Buch von Daniel Wollenschläger (2017). Grundlagen der Datenanalyse mit R. Springer Spektrum, Berlin Heidelberg (Buch als pdf für Angehörige einiger Hamburger Universitäten).
Tipps und Tricks zum Abspeichern von Tabellen
Um eine Tabelle mit Ergebnissen aus R zu exportieren, wird die Funktion write.table() verwendet.
Eine sehr beliebte Funktion zur Berechnung von deskriptiver Statistik ist die summary() Funktion.
Sie gibt die Parameter Mittelwert, Median, Minimal- und Maximalwert, sowie die 1. und 3. Quartile in einem einzigen Schritt aus.
deskript_stats <- summary(iris)
> deskript_stats
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100 setosa :50
1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300 versicolor:50
Median :5.800 Median :3.000 Median :4.350 Median :1.300 virginica :50
Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500 Obwohl hier ein Dataframe zugewiesen wurde, lässt sich dieser nicht ohne Weiteres exportieren. Exportieren natürlich schon, allerdings nicht in einer ansehnlichen Formatierung:
write.csv(deskript_stats, "G:/Rlab/deskriptive_statistik_iris.csv", dec=".", sep=";") Wird die Datei in Excel geöffnet, stehen in jeder Zelle auch noch die statistischen Kenngrößen. Hier müsste nun ,,per Hand" die Tabelle berichtigt werden.
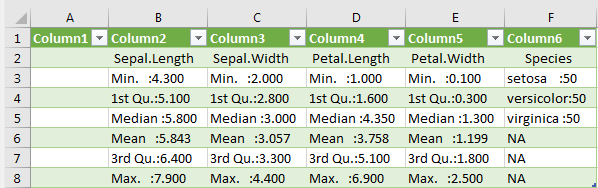
Wie geht es einfacher?
Mit der Kombination aus cbind und lapply werden die Zeilenbeschriftungen in einer Spalte zusammengefasst und die deskriptiven Statistikparameter für jede Spalte einzeln berechnet.
So lässt sich dann der Dataframe wie gewohnt exportieren und die .csv Datei kann für weitere Verwendungen benutzt werden.
deskript_stats_1 <- do.call(cbind, lapply(iris, summary))
> deskript_stats_1
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
Min. 4.300000 2.000000 1.000 0.100000 50
1st Qu. 5.100000 2.800000 1.600 0.300000 50
Median 5.800000 3.000000 4.350 1.300000 50
Mean 5.843333 3.057333 3.758 1.199333 50
3rd Qu. 6.400000 3.300000 5.100 1.800000 50
Max. 7.900000 4.400000 6.900 2.500000 50
write.csv(deskript_stats_1, "G:/Rlab/summary_iris.csv", dec=".", sep=";")Wird die .csv Datei nun in Excel geöffnet, stehen deskriptiven Statistikparameter in einer Spalte untereinander und die Werte jeweils in einer Zelle.
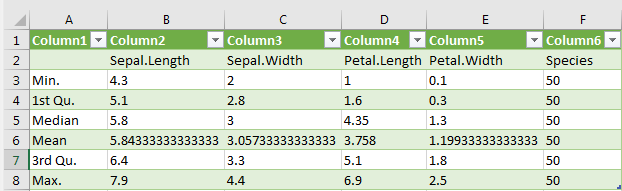
Weitere nützliche Hinweise findest Du auch im Skript zu ,,Export von Daten in R" unter Wie exportiere ich Daten im Comma-Speparated-Values-Format?.
Weitere nützliche Hinweise findest Du auch im Skript zu ,,Import von Daten in R" unter Importieren von Comma-Seperated-Values- und Text-Dateien in R.
Nützliche Funktionen, mit denen Du überprüfen kannst, ob Deine Daten richtig in R importiert wurden findest Du auch im Skript zu ,,Import von Daten in R" unter Wurden meine Daten korrekt in R importiert?.
Die Kunst des Skriptens: Namen in R
Bevor Du Dein R-Skript erstellst, sind im Folgenden einige nützliche Hinweise zusammengestellt:
Beim Erstellen eines Skriptes (Name.R) sollte besondere Sorgfalt beim Vergeben der Objektnamen aufgewendet werden.
Es gibt einige ,,ungeschriebene" Gesetze, die Du beachten solltest. Ein R-Skript sollte logisch, gut dokumentiert und strukturiert aufgebaut sein.
So kannst nicht nur Du Dich an Deine Analyseschritte auch nach einiger Zeit noch erinnern, sondern auch andere können Deine Skripte nachvollziehen und Dir gegebenenfalls helfen.
Skript-Dateiname.R
In Skript-Dateinamen (Name.R) sind keine Umlaute erlaubt.
Ebenso sollten keine !, +, - , #, *, verwendet werden.
GOOD: fit-models.R („sprechender” Name)
BAD: stuff.R
Variablennamen
Variablennamen sollten kleingeschrieben werden und Worte sollten durch einen Punkt "." oder einen Unterstrich "_" unterteilt werden.
GOOD: avg_clicks („sprechender” Name)
OK: avgClicks
BAD: ac
Kurze, prägnante Namen sind immer empfehlenswerter als lange Namen.
GOOD: desk_stat_df_1
OK: deskript_stat_df_1
BAD: deskriptive_statistik_von_dataframe_1
Eindeutige Objektbezeichnungen sind empfehlenswert. Werden Objekte gleich benannt, so werden diese überschrieben und es bei Berechnungen zu Fehlern kommen, da nicht auf das ,,gewünschte" Objekt zugegriffen wird.
Namen, die folgende Symbole beinhalten sollten vermieden werden:
?, $, %, ^, &, *, (, ), -, #, ?, ,, <, >, /, |, \, [, ], {, und }
Weitere nützliche Hinweise zum Thema ,,Objekt- und Spaltenbezeichnungen" findest Du auch im Skript zu ,,Import von Daten in R" unter Vorbereiten von Daten.
Die Kunst des Skriptens: Formatierung des Skripts
aus: Crockford, D. (2008): JavaScript: The Good Parts. Yahoo Press / O'Reilly. Sebastopol, USA."It turns out that style matters in programming for the same reason that it matters in writing. It makes for better reading.“
Warum sind Richtlinien zur Formatierung des Skripts (sog. ,,Styleguide") wichtig?
,,Es gibt nur einen Grund, sich an einen Styleguide zu halten: Wir wollen, dass unser Quellcode nicht nur von einem Computer, sondern auch von einem Menschen interpretierbar ist."
Ein Programmierstil legt zum Beispiel fest, wo Leerzeichen gesetzt werden oder wie Datei- und Objektnamen aufgebaut sind. Ein einheitlicher Stil hat große Vorteile, aber keinerlei Auswirkungen auf die Funktionalität des R-Codes. Fehler lassen sich deutlich einfacher finden und die Lesbarkeit, insbesondere wenn mehrere Personen am Code arbeiten, wird erhöht.
Vorteile der Lesbarkeit des R-Codes:
- Deine Kollegen werden Dich mögen und machen Deine Arbeit gerne dort weiter, wo Du aufgehört hast. Zumindestens könnt ihr das so leichter erreichen. ;)
- Fehler sind leichter zu erkennen.
- Wenn Du Deinen eigenen Quellcode überprüfst, wirst Du Dich seltener fragen: Wer hat das geschrieben? Und was bedeutet das?
- Wenn Du Deinen eigenen Quellcode nach einiger Zeit wieder verwenden willst, wirst Du Dich seltener fragen: Wer hat das geschrieben? Und was bedeutet das?
Styleguide-Empfehlungen
Einen sehr empfehlenswerten Styleguide gibt es auf der Advanced R-Webseite, einem Zusatzangebot zu einem gleichnamigen Buch.
Weitere:
Beachte: Der Programmierstil hat keinerlei Auswirkungen auf die Funktionalität! Es geht nur um Lesbarkeit.
Empfehlungen: Leerzeichen
Binäre Operatoren =, +, -, <- sollten von Leerzeichen umschlossen sein.
Es sollte kein Leerzeichen vor einem Komma gesetzt werden, sondern immer danach.
Aber: Leerzeichen beeinflussen die Verarbeitung des Codes durch R nicht!
Und, wie später ausführlicher erwähnt, nicht vergessen: Dokumentation der einzelnen Schritte im Skript --> LIVESAVER ! Hinter dem #-Zeichen ausführlich, später nachvollziehbar notieren, was die Funktion macht und wofür einzelne Argumente da sind!
Die Kunst des Skriptens: Groß- und Kleinschreibung
Im Gegensatz zu vielen anderen Programmiersprachen ist R ,,case sensitive". Das bedeutet, dass große und kleine Buchstaben unterschiedlich interpretiert werden. Zum Beispiel ist x ist nicht das gleiche wie X oder Objekt1 nicht objekt1. Diese Tatsache ist enorm wichtig zu beachten, da Objekte und Funktionen, wenn die Groß-/Kleinschreibung nicht stimmt, nicht gefunden werden können. Dies ist eine häufige Ursache für Fehler, die sich leicht vermeiden lässt.Erschwerend kommt hinzu, dass sich einige Zeichen in der ,,R-Schriftart" schlecht unterscheiden lassen, zum Beispiel:
- l und 1 (Kleinbuchstabe l und Ziffer 1)
- 0 und O (Ziffer Null und Großbuchstabe O)
- v und V (Klein- und Großbuchstabe V)
- x und X (Klein- und Großbuchstabe X)
Das klingt trivial, führt aber oft zu schwer auffind- und lösbaren Fehlern, da der R-Code auf den ersten (und häufig auch zweiten, dritten,...) Blick fehlerfrei aussieht.
Das perfekte R-Skript
Beginn eines R-Skripts
Es empfiehlt sich, zu Beginn es R-Sktiptes, immer eine ,,Kopfzeile" einzufügen.
Diese könnte wie folgt aussehen:
###################################################################
# R-CODE FÜR "HIER DEN TITEL DES R-SKRIPTES EINFÜGEN"
# "DATUM EINFÜGEN"
# "AUTOR ODER URSPRUNGS_URL EINFÜGEN"
###################################################################
rm(list=ls()) # hier wird alles aus dem Environment gelöscht
# wichtig, da alte Dateien die folgenden Berechnungen stören könnten
# # -----------------------------------------------------------------------
# Working Directory setzen
setwd("PFAD_EINFÜGEN")
# Packages laden
library(ggplot2)
# Mein erstes Skript ------------------------------------------------------
# Soil-Daten einlesen
soil <- read.table("PFAD_EINFÜGEN", header=T, sep=";", dec=".")
# Meine ersten Berechnungen -----------------------------------------------
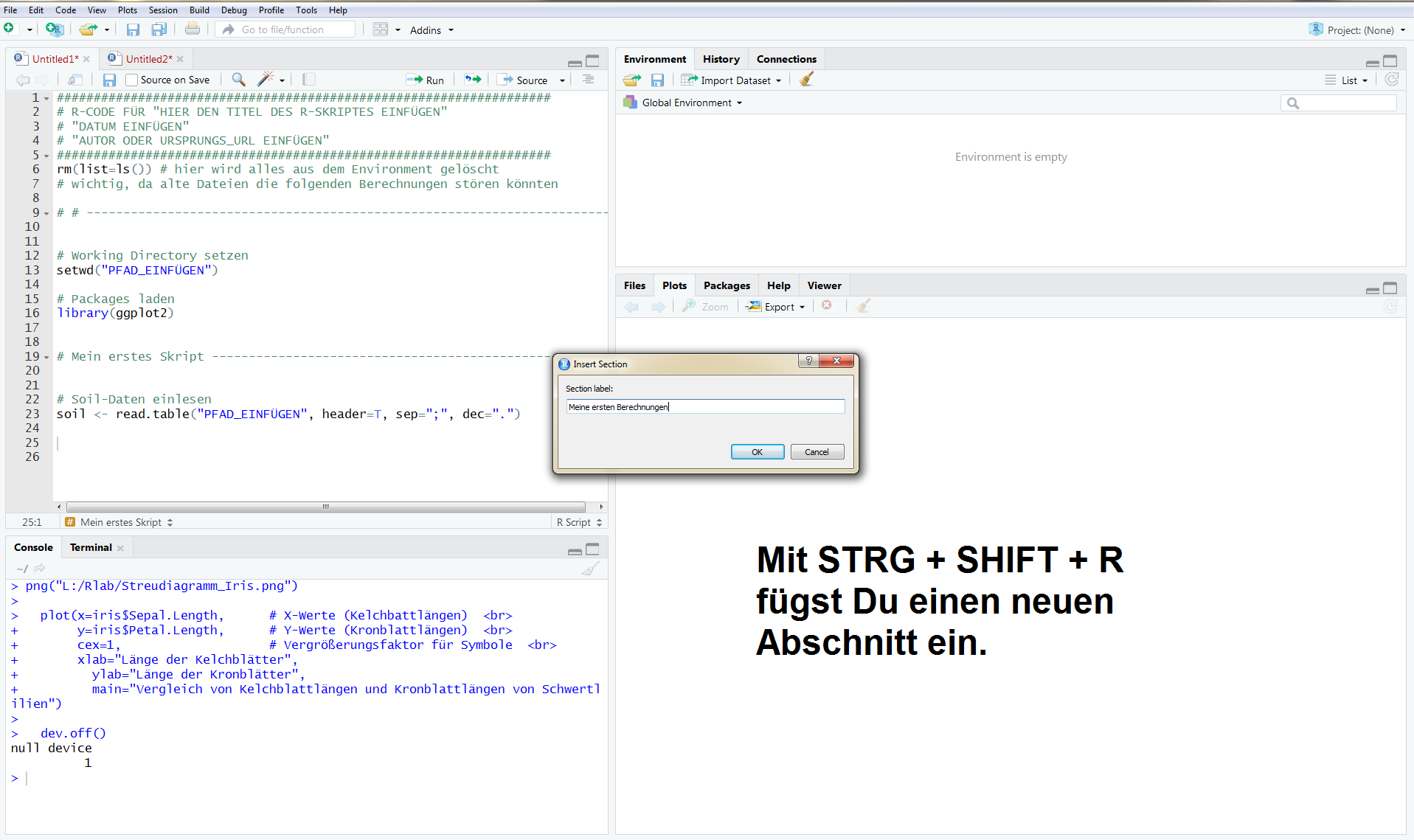
In der folgenden Abbildung siehst Du wie das ganze in R aussieht.
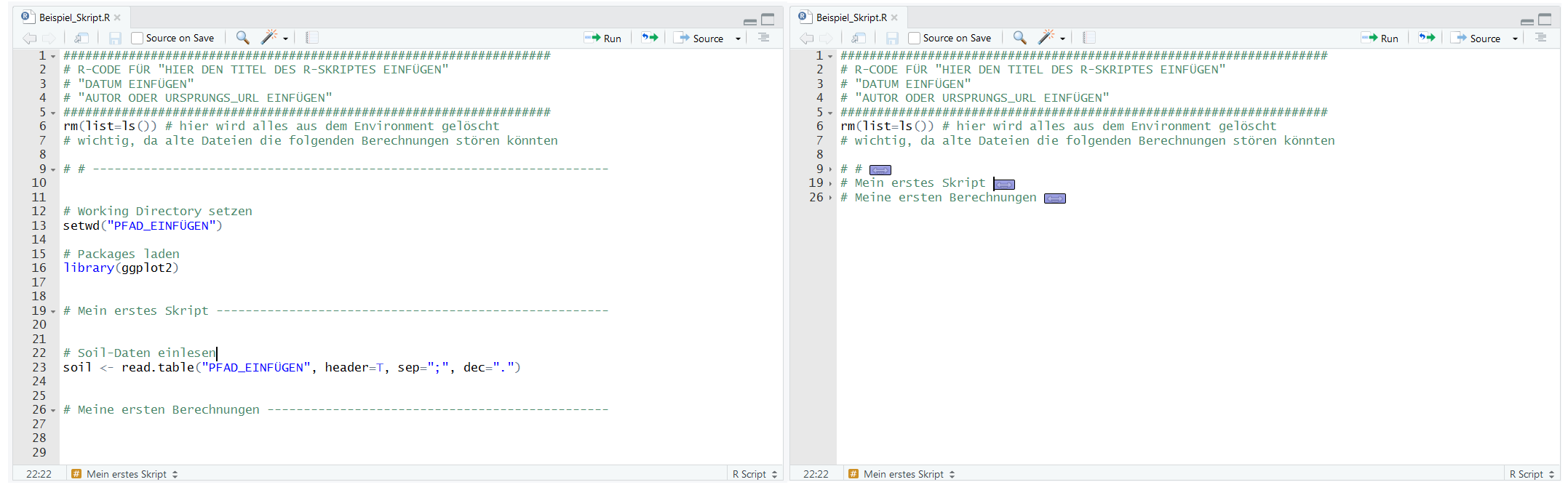
Die Verwendung von mindestens ##### oder ---- oder ==== ermöglicht, dass ein neuer Abschnitt im Skript eingefügt wird.
Es erscheint an der Seite des Skriptes ein kleiner Pfeil, mit dem der in diesem Abschnitt enthaltenden Code ,,einklappen" kannst. Dadurch kann schon bearbeiteter oder nicht mehr benötigter Code ,,ausgeblendet" werden und das Skript ist übersichtlicher.
Folgende Tastenkürzel ermöglichen Dir Sektionen in Dein Skript einzubauen und zu verwalten.
- Neuen Abschnitt einfügen — Ctrl+Shift+R (Cmd+Shift+R auf dem Mac)
- Zum nächsten Abschnitt springen — Shift+Alt+J
- Aktuellen Abschnitt einklappen — Alt+L
- Aktuellen Abschnitt ausklappen— Shift+Alt+L
- Alle Abschnitte einklappen — Alt+O
- Alle Abschnitte ausklappen— Shift+Alt+O
Mit STRG + SHIFT + R erscheint ein neues Fenster, in dem Du den Abschnittsnamen vergeben kannst.
Hier siehst du die ,,ausgeklappten" und ,,eingeklappten" Abschnitte im Vergleich
Die Kommentar-Funktionen
Im Vergleich zu anderen Statistiksoftwares wie Excel, SPSS oder SAS, hat R einen entscheidenden Vorteil: die Kommentar-Funktion.
Mit dem # Symbol kannst Du zu jedem Schritt einen Kommentar schreiben und so Deinen Lernprozess dokumentieren. Deine Versuche sind also keinesfalls umsonst oder verschwendete Zeit, sondern ganz einfach ein Lernprozess.
Nur durch das eigenständige Ausprobieren, Kombinieren und der Fähigkeit ,,wie das Programm zu denken“ wirst Du R lernen.
Ehe Du Dich versiehst, wird sich der Umwang mit R zu einer Selbstverständlichkeit entwickeln.
Die Dokumentation der einzelnen Schritte in einem Skript sind Deine Lebensversicherung ! Dokumentiere möglichst genau Deine einzelnen Schritte und auch die Ergebnisse.
Weitere nützliche Hinweise findest Du auch im Skript zu ,,RStudio und R-Skripte" unter Vorteile beim Arbeiten mit R-Skripten und im Skript zu ,,Import von Daten in R" unter Vorbereitung von Daten.
Fragen oder Anmerkungen?
Für Anregungen und Kommentare zur Verbesserung dieses Digitalen Skripts ist das RLab-Team immer dankbar! Du kannst auch Fragen zu den Inhalten stellen! Nutze für all das gerne die Kommentar-Funktion!
Die R Cheat Sheets
Da es ist unmöglich ist sich alle Funktionen zu merken, muss man nur wissen wo man sich die entsprechenden Informationen besorgen kann!.
Das R ,,Cheat Sheet R Base" bietet eine gute Übersicht zu den wesentlichen Funktionen und Argumenten des bereits in R implementierten Package base.
Gerade dieses Cheat Sheet ist sehr hilfreich bei den Grundfunktionalitäten und Arbeitsweisen von Vektoren und anderen Datenformaten, Schleifen, mathematischen Funktionen und Tests R Cheat Sheet Base.
Ebenso empfehlenswert sind die Cheat Sheets zu Plot-Darstellungen. Eine gute Übersicht zu den Gundfunktionen findest Du im R Base Graphics Cheat Sheet, alles zu Formatierungen der graphsichen Darstellung findest Du im R Base Cheat Sheet Graph Sizes.
Auf der Internetseite von RStudio sind viele weitere R Cheat Sheets, getrennt nach Packages, zusammengestellt. Hier gelangst Du zu den kostenlosen Downloads.Literatur
Zusätzlich zu den in diesem Digitalen Skript zitierten und für die Erstellung verwendeten Quellen sind einige weitere Ressourcen angegeben, die den Einstieg und die Arbeit mit R erleichtern können.
- Dormann, C.F. 2013. Parametrische Statistik. Springer Spektrum, Berlin Heidelberg (Buch als pdf für Angehörige einiger Hamburger Universitäten)
- Hatzinger, R., Hornik, K., Nagel, H., Maier, M.J. 2014. R: Einführung durch angewandte Statistik. Pearson, Hallbergmoos (Buch als pdf für Angehörige der Universität Hamburg)
- Wollschläger, D. 2017. Grundlagen der Datenanalyse mit R. Springer Spektrum, Berlin Heidelberg (Buch als pdf für Angehörige einiger Hamburger Universitäten)
- Wollschläger, D. 2016. R kompakt: der schnelle Einstieg in die Datenanalyse. Springer Spektrum, Berlin Heidelberg (Buch als pdf für Angehörige einiger Hamburger Universitäten)
- Wollschläger, D. R Examples Repository RExRepos [sehr guter Beispielcode für häufig benötigte Standardrechnungen, englischsprachig, letzter Aufruf 23.04.2019]
- Zuur, A. et al. 2009. A beginner's guide to R. Springer, New York (Buch als pdf für Angehörige einiger Hamburger Universitäten)
Zusammenfassung
In diesem Digitalen Skript wurden Dir Tipps und Tricks gezeigt, die Dir das Erlernen und den Umgang mit R erleichtern sollen.
Weitere Hinweise findest Du in den themenspezifischen Digitalen Skripten.
Fragen oder Anmerkungen?
Für Anregungen und Kommentare zur Verbesserung dieses Digitalen Skripts ist das RLab-Team immer dankbar! Du kannst auch Fragen zu den Inhalten stellen! Nutze für all das gerne die Kommentar-Funktion!
Dir gefällt RLab?
Nimm Dir ein R ! Alles ist besser mit einem R!